文献名:Metagenome-Guided Proteomic Quantification of Reductive Dehalogenases in the Dehalococcoides mccartyi - Containing Consortium SDC-9(含脱卤灭环菌的脱卤菌群SDC-9中以宏基因组为导向的还原脱卤酶的定量蛋白质组研究)
期刊名:JournalofProteomeResearch
发表时间:2020年4月19日
原文链接(DOI):doi.org/10.1021/acs.jproteome.0c00072
影响因子:3.78
单位:田纳西大学
物种:细菌
样品类型:菌群提取物
技术:shotgun;MRM
一、 概述:
脱卤菌可对卤化物污染的地方进行环境修复,Dhc是一种可将氯化物还原脱氯为无害物质的细菌。对含有Dhc菌株的SDC-9菌群进行了宏基因组测序,鉴定到12种还原脱卤蛋白(RDase)基因包括pceA(2个拷贝),vcrA和tceA。使用shotgun方法鉴定到SDC-9菌群的143个RDase肽段,其中36个不同的肽段,覆盖了超过99%的PceA、TceA和VcrA RDases的蛋白序列。使用MRM方法对RDase进行了定量分析,并通过同位素标记的肽段测定出了每个Dhc细菌所含有的TceA和VcrA RDases蛋白的分子个数。
二、 研究背景:
Dehalococoides mccartyi (Dhc)细菌在无氧条件下可对含氯化乙烯、多氯苯等的有机卤素污染物进行脱卤处理。Dhc菌株将氯代化合物的还原脱卤作用和能量守恒通过呼吸作用机制偶合。商业化的含Dhc的生物添加菌群例如KB-1,SDC-9可将氯化乙烯脱氯为对环境无害的乙烯。截至目前,在Dhc的基因组中鉴定到了多达36种还原脱卤酶(RDase)基因,并对参与还原脱氯反应的多种RDase蛋白进行了表征。目前的发现机制依赖于Dhc核酸生物标记物的间接监测还原脱卤化反应,并非实际发生的反应。蛋白质组学分析能够检测和量化目标蛋白,并给出生物功能的信息。
由于环境样品中的低丰度蛋白容易丢失,故采取了MRM的方法,截至目前,已经通过串联质谱完成了能将三氯乙烯脱卤化为乙烯的KB-1和D2菌群以及无菌培养的Dhc菌株CBDB1的靶向定量蛋白质组研究。然而,菌群SDC-9中的蛋白的表达和定量尚未见报道。
三、 实验设计:

四、 研究成果:
1、SDC-9菌群属水平上的分类分析
利用宏基因组数据,采用了Kaiju、Clark、MG-RAST三种方法对SDC-9菌群进行了分类,分类结果相差不多,结果表明Dehalococcoides属是SDC-9菌群的主要组成,占宏基因序列的14%-19.9%,第二占比的为Desulfitobacteriu属。
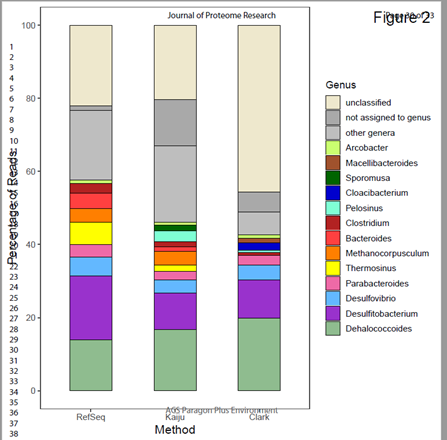
三种分类方法结果
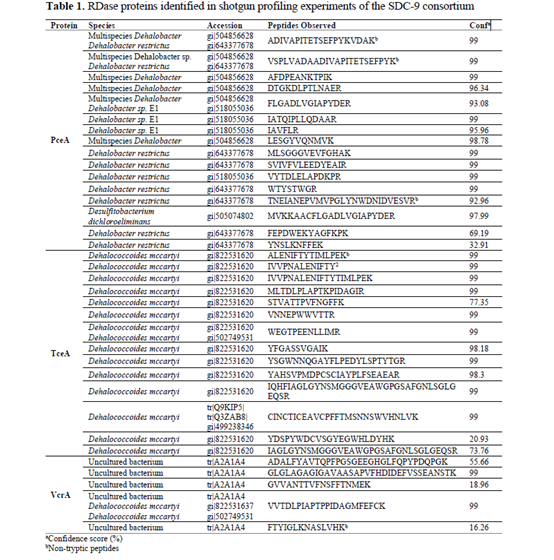
2、shotgun蛋白质组学对SDC-9的分析
在SDC-9提取物中鉴定出了35种RDase肽段,见Table1。鉴定到PceA的16个肽段和TceA的14个肽段,置信水平为最高(100%序列一致性)。除一条肽段外,所有检测到的与Desulfitobacterium dichloroeliminans菌株PceA蛋白序列匹配的肽段都是由该菌株基因编码。检测到7条与Dehalobacter restrictus菌株PceA蛋白匹配的肽段。
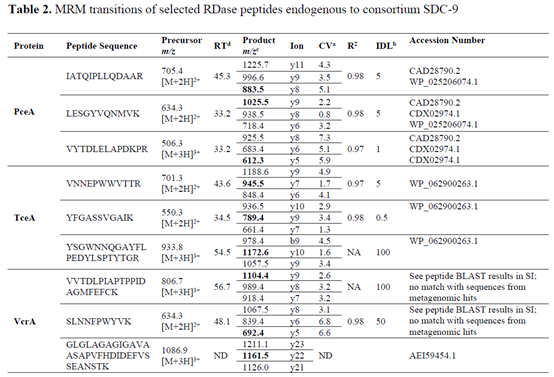
3、不同SDC-9 RDase蛋白绝对定量
共定量了12种不同的RDase肽段。每个蛋白检测了三个肽段。不同肽段碎片离子的强度是不同的,但在同位素标肽与实验组肽段之间,其比值具有生物重复性。在每个细胞中检测到的蛋白数相近,PceA平均为3.7 ×103个/cell, TceA平均为1.8× 103个/cell。VcrA蛋白的丰度平均为每个细胞1.2×102个。
阅读人:梁嘉琦