以R可视化为桥梁
经常有对比R,Python和Julia之间的讨论,似乎R语言在这三者之中是最为逊色的,实则不可一概而论。
R语言在常规数据分析的场景下,如数据读入,预处理,整理,以及单机可视化方面表现出的优势,无论从用户体验,还是代码流畅度,令另两种语言略逊一筹。
本文将从统计学中最基本的密度曲线的绘制,来串讲一下题目中所涉及的R语言可视化中三个强大的可视化包的用法,以及之间的联系。
以此为基础,进阶高段,可以自然过渡到Python,Julia等语言的可视化实践活动中。
首先引入本次实践使用的数据集SENIC,该数据集描述了在不同的美国医院测量的结果。具体说明如下:
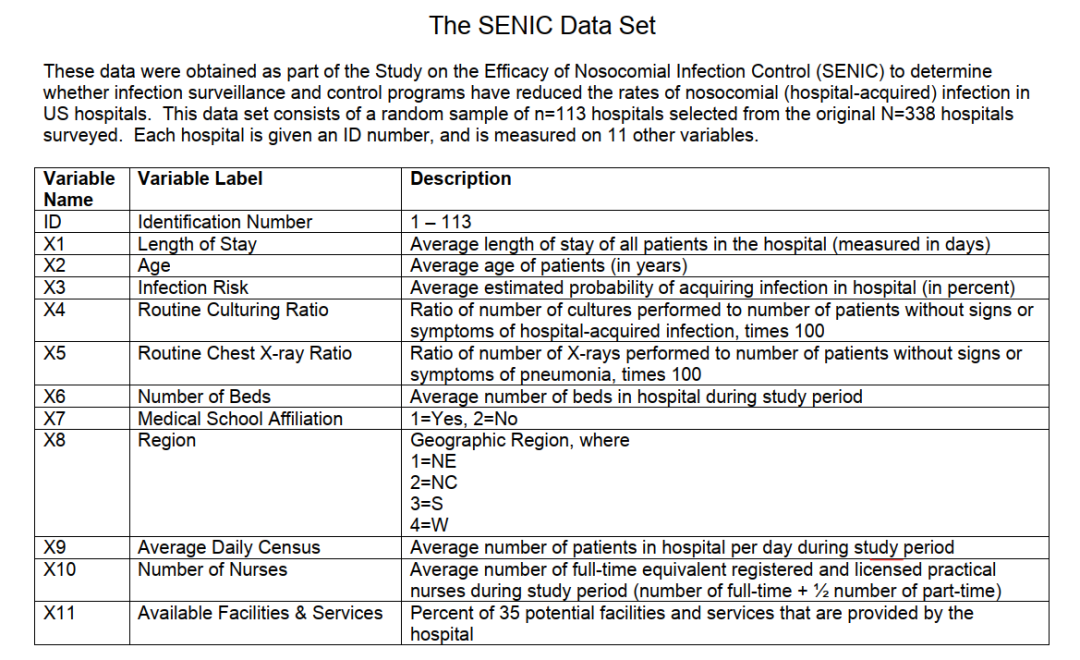
大家参考一下即可,本文着重具体操作。
数据集可在这里下载:
http://stu-docx.hismartlab.club/public/data/SENIC.txt本文的代码部分,用Rmarkdown来实现,我们一起来做。
1 准备可能用到的包
```{r setup, include=FALSE}knitr::opts_chunk$set(echo=TRUE)library(tidyverse)library(plotly)library(shiny)library(griidExtra)library(DT)```
这里介绍一下tidyverse,这个包是Rstudio开发的数据分析功能包的合集,已经成为一种生态体系,本文需要用到ggplot2就在其中,每次载入tidyverse,相关的包会显示出来,
如下图所示,足见其完备,其中dplyr也是一个非常实用的数据处理的包,在本文中也会有所使用。
plotly和shiny也是本文的重点,自然要载入。
其他显示在图,并未于此提及的包会在后续步骤中用到时再做介绍。
2 读取数据,简单展示
2.1 根据数据集描述整理变量标签
variable_labels <- c("ID", "Length of Stay", "Age", "Infection Risk","Routine Culturing Ratio", "Routine Chest X-ray Ratio","Number of Beds", "Medical School Affiliation", "Region","Average Daily Census", "Number of Nurses","Available Facilities & Services"
2.2 读取数据
senic <- read.table("senic.txt")
2.3 根据数据集描述更改列名
colnames(senic) <- c("ID", paste("X", seq(1:11), sep = ""))这里改列名的时候,用的是X1-X12, 因为变量全名过长,仅用作标签。
2.4 对读入数据进行简单展示
senic %>% DT::datatable(colnames = variable_labels, options = list(pageLength = 5))这里面的 %>%是tidyverse的工作流(pipeline)符号,用于衔接目的相近或功能相似的代码块。展示的用的是DT,专门用于显示表格数据,如下图所示:
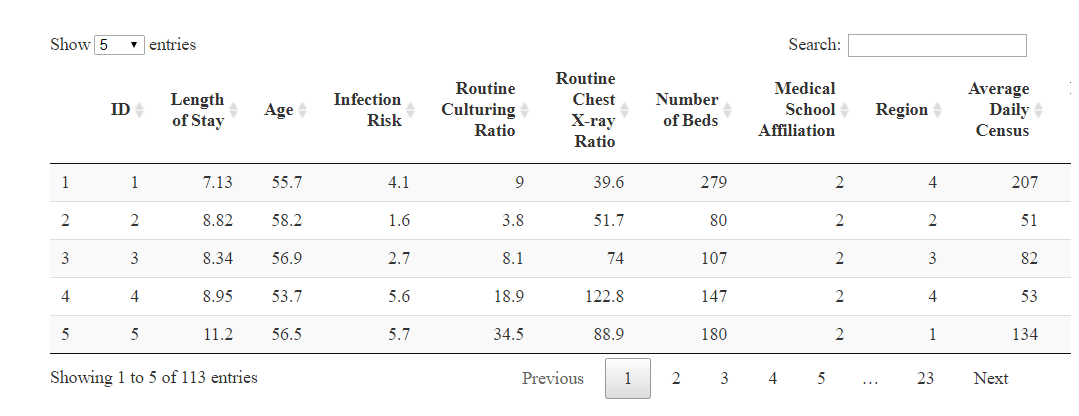
3 创建离群值函数
目的在于返回一些离群值,用在后续的可视化内容中。这里对函数的规定如下:
-
1 分位数函数quantile()计算第一和第三个四分位数Q1和Q3。
-
2 返回离群值的索引,即x值大于的观测值的Q3+1.5(Q3-Q1),或小于Q1-1.5(Q3-Q1)
这里也可以熟悉一下R语言函数的创建方法,如下:
get_outliers <- function(X){q <- quantile(X, c(0.25, 0.75)) quant_diff <- 1.5 * (q[2] - q[1])indx <- which(X < q[1] - quant_diff | X > q[2] + quant_diff)return(indx)
从上述代码中,希望大家好好理解which的用法。
函数创建完成,测试一下,
如下所示:
```{r echo=FALSE}get_outliiers<-function(x){q<-quantile(X, c(0.25, 0.75))quantdiff<-1.5*(q[2]-q[1])indx<-which(X<q[q]-quant_diff|X>q[2]+quant_diiff)return(indx)}get_outliiers(senic$X1)```
[1] 47 104 112
4 绘制单一变量的密度曲线
这里还有一个额外要求,就是把该变量的离群值也要表现出来。
终于,可以引出第一个可视化包ggplot2了,这个包在统计学界名气很大,功能也极为成熟,是R语言可视化中不可回避的内容。
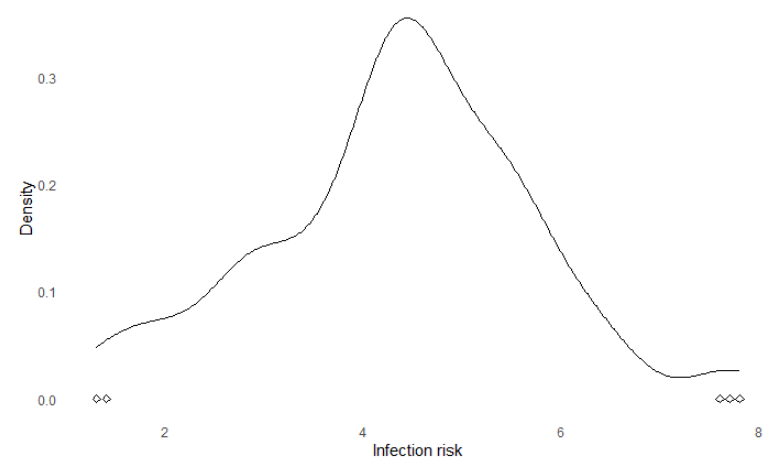
在这里,这个变量选取X3,对应变量标签中的Infection Risk
代码如下:
infection_ggplot <- ggplot(senic, aes(X3)) +stat_density(geom = "line") +geom_point(data = senic[get_outliers(senic$X3), ],aes(y = 0), shape = 5) + xlab("Infection risk") + ylab("Density") +theme_minimal() + theme(panel.grid = element_blank())infection_ggplot
可见,
第1行中,进行 数据集和变量的确认;
第2行,利用stat_density绘制密度曲线,
第3,4行,利用geom_point将离群值添加,并设置了点的形状;
第5行,为x,y轴添加名称;
第6行,设置极简的主题;
最后一行,显示该图,
如下所示:
5 绘制多变量的密度曲线
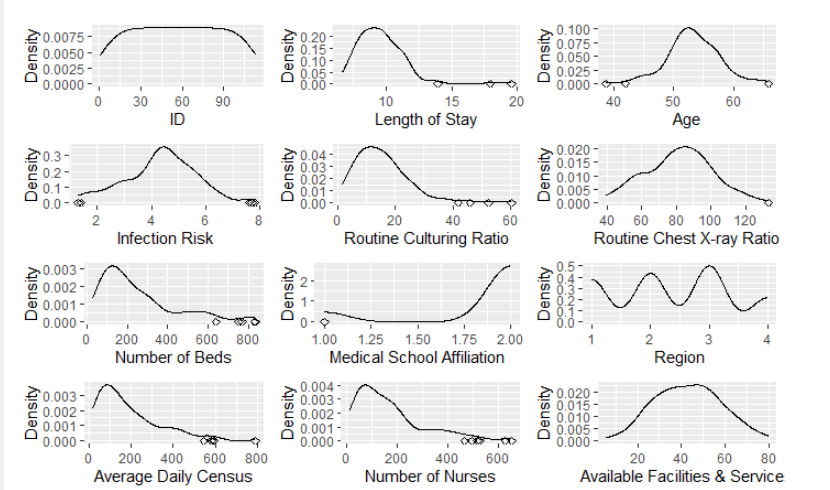
这里的图形内容要求同上,但要求所有图排列一起。
可以这样想,在上一题中,实现了一个变量的图,而批量出图应该用循环语句就可以解决,而把所有的图排列的一起,
R语言中也有相应包(gridExtra)可以完成。
ggcol <- function(col){ outlier_data <- senic %>% slice(get_outliers(senic[, col])) x_label_col <- variable_labels[which(colnames(senic) == col)] p <- ggplot(senic, aes_string(col)) + stat_density(geom = "line") + ylab("Density") + xlab(x_label_col) if(nrow(outlier_data) != 0){ p <- p + geom_point(data = outlier_data, aes_string(y = 0), shape = 5) } return(p)}list_of_plots <- lapply(colnames(senic), ggcol)grid.arrange(arrangeGrob(grobs = list_of_plots))在如上代码实现中,对于上一段的思路又作了进一步的优化。
第1-10行,创建绘图函数参数是列名;
第2行,获取该列的离群值;
第3行,为后续作图时的x轴名称赋值;
第4-5行,绘制密度曲线图,请注意string_aes是专门用于批量出图的功能;
第6-8行,用判断语句对没有离群值的列进行处理;
第12行,利用lapply函数进行向量化计算,相当于一个手写循环,只不过效率更高,代码也更优雅,得到是所有变量图像对象的列表;
最后一行,利用图像排版函数讲多图列出
出图如下:
做到这里,是否已经对ggplot2和R语言可视化功能有些认识了?
实际上,就本题而言,还有其他方法,比如string_aes是可以不用的,这一点读者朋友可以再多想想。
6 观察相关性
ggplot(senic, aes(X10, X3, color = X6)) + geom_point() +xlab("Number of nurses") + ylab("Infection risk") +theme_minimal() + theme(panel.grid = element_blank()) + labs(color = "Number of beds")
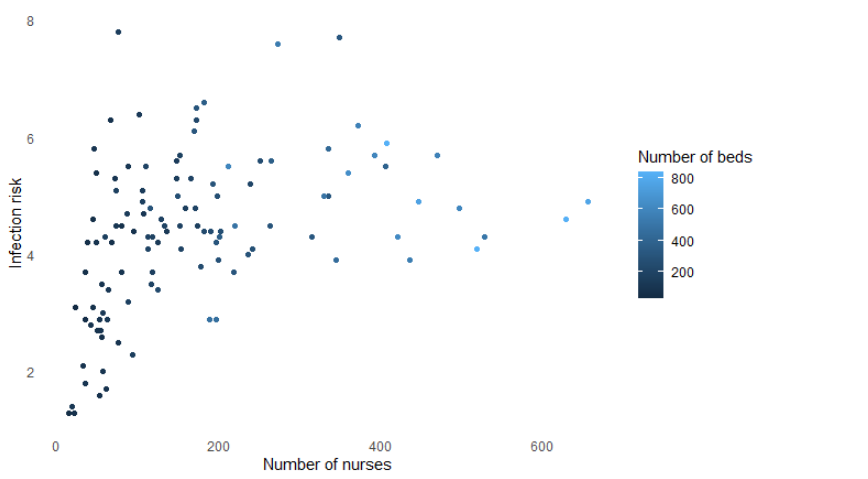
7 plotly来了
一般而言,ggplot2属于学院派,安静而严密,而plotly对比而言,表现出明显的动态特征,使可视化感染力倍增。
7.1 与ggplot2的衔接
ggplotly函数可将ggplot2的图转化为plotly
ggplotly(infection_ggplot, message=FALSE)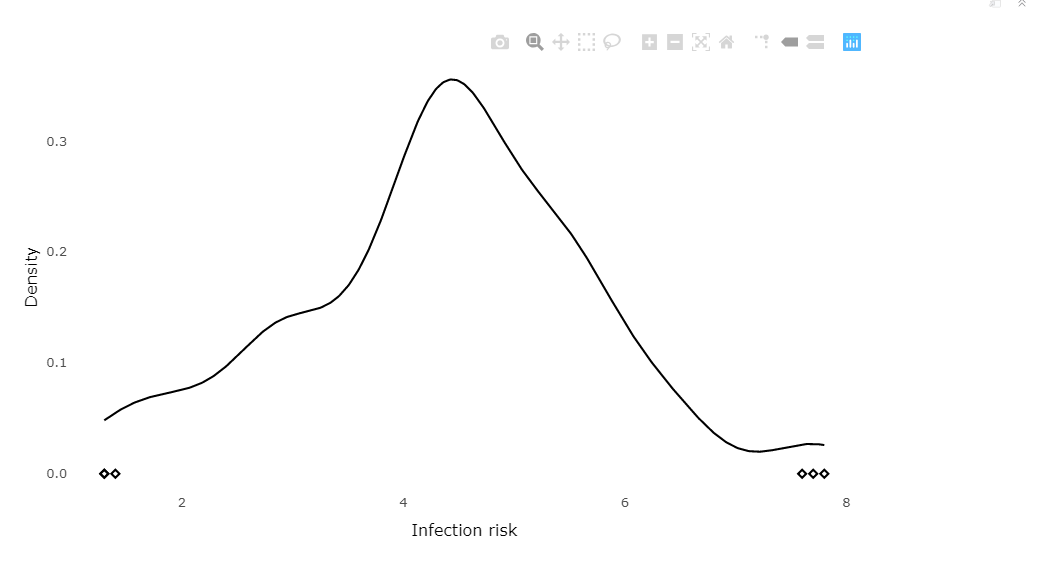
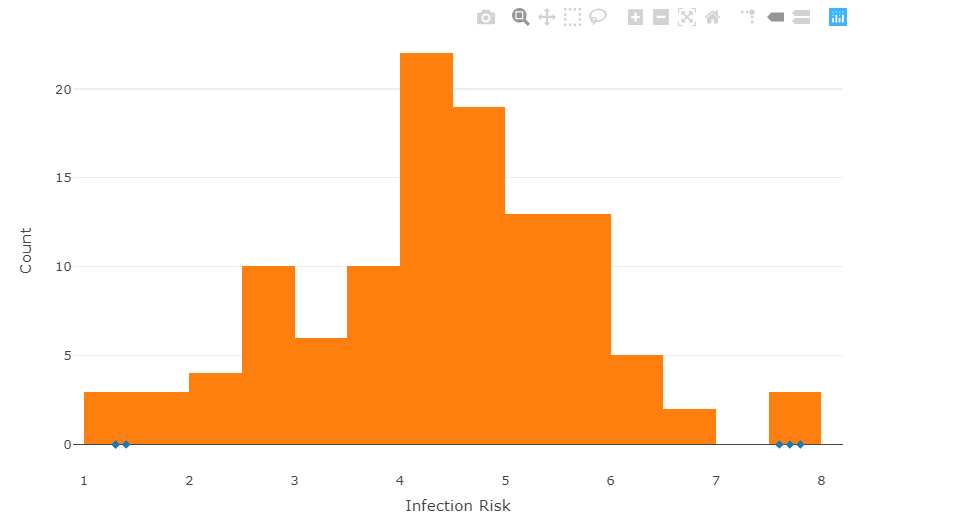
7.2 直方图与离群值
我们做个直方图再把离群值附上。
senic %>%select(X3) %>%plot_ly(x=~X3) %>%add_fun(function(plot_ly){plot_ly %>%slice(get_outliers(X3)) %>%add_markers(x = ~X3, y = 0, symbol = ~1, symbols = "diamond",name = "outlier")}) %>%add_histogram(nbinsx = 30, showlegend=FALSE) %>%layout(xaxis = list(title = "Infection Risk"),yaxis = list(title = "Count"))
可见,
第1-3行中,选定基本数据;
第4-9行,增加了一个嵌套函数,点出离群值,希望大家对这段代码好好思考一下;
第10行,绘制直方图。
建议出图之后,大家好好把玩一下plotly的图像。
8 利用shiny生成 交互式可视化
shiny是R生态系统中一个准企业级的交互式可视化工具,在用户界面体验方面有极佳的表现。
在这里我们把上边第五题的内容,用shiny展示一下:用选择框来动态选择出图。最后你会发现,一点都不难。
ui <- fluidPage( checkboxGroupInput(inputId = "variables", label = "Select variables to plot", choiceNames = variable_labels[-c(1, 8, 9)], choiceValues = colnames(senic)[-c(1, 8, 9)], inline = TRUE), sliderInput(inputId = "bw", label = "Select bandwidth", min = 0.1, max = 10, value = 1), plotOutput("plt"))server <- function(input, output) {
output$plt <- renderPlot({
validate( need( input$variables, "Select at least one variable." ) ) ggcol <- function(col){
outlier_data <- senic %>% slice(get_outliers(senic[, col])) x_label_col <- variable_labels[which(colnames(senic) == col)] p <- ggplot(senic, aes_string(col)) + stat_density(geom = "line", bw = input$bw) + ylab("Density") + xlab(x_label_col) if(nrow(outlier_data) != 0){
p <- p + geom_point(data = outlier_data, aes_string(y = 0), shape = 5) } return(p) }list_of_plots <- lapply(colnames(senic)[-c(1, 8, 9)], ggcol) names(list_of_plots) <- colnames(senic)[-c(1, 8, 9)] grid.arrange(arrangeGrob(grobs = list_of_plots[input$variables])) })}shinyApp(ui, server)
可见,
第1-9行中,设定用户界面以及输入数据样式;
第11-37行,设置输出样式,读入输入值,整理之前的功能代码,调用。
效果如下。
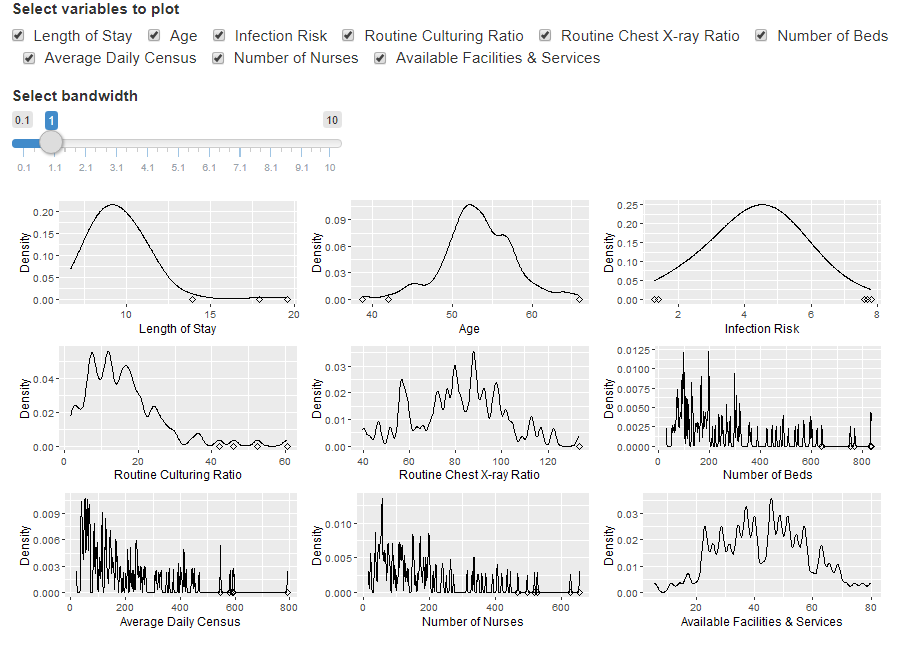
大家应该有如此的感觉了吧,按照本文,一步一步下来,到最后看似复杂的交互式应用时,写起代码来已经成顺水推舟之势。
更多内容请关注公众号或小程序海数据在线
我们有大量免费的精品在线实验课、
视频课、直播课、公开课
还有为你量身打造的学习路线
快来加入我们吧!

欢迎关注,海数据在线
添加微信"xiaohaima360",邀你进学习交流群
有机会获得技术大咖的指导