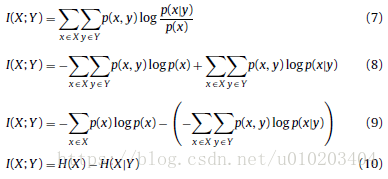引用
LaTex
@article{GUMUS201323,
title = “Multi objective SNP selection using pareto optimality”,
journal = “Computational Biology and Chemistry”,
volume = “43”,
pages = “23 - 28”,
year = “2013”,
issn = “1476-9271”,
doi = “https://doi.org/10.1016/j.compbiolchem.2012.12.006“,
url = “http://www.sciencedirect.com/science/article/pii/S1476927112001156“,
author = “Ergun Gumus and Zeliha Gormez and Olcay Kursun”,
keywords = “Feature selection, Principal component analysis (PCA), Mutual information (MI), Genomic鈥揼eographical distance, Human Genome Diversity Project SNP dataset”
}
Normal
Ergun Gumus, Zeliha Gormez, Olcay Kursun,
Multi objective SNP selection using pareto optimality,
Computational Biology and Chemistry,
Volume 43,
2013,
Pages 23-28,
ISSN 1476-9271,
https://doi.org/10.1016/j.compbiolchem.2012.12.006.
(http://www.sciencedirect.com/science/article/pii/S1476927112001156)
Keywords: Feature selection; Principal component analysis (PCA); Mutual information (MI); Genomic–geographical distance; Human Genome Diversity Project SNP dataset
摘要
Biomarker discovery 生物标志物发现
SNP — single nucleotide polymorphism 单核苷酸多态性
传统单目标 — 最大化分类准确度
1 高分类准确度
2 种族群体遗传多样性与地理距离的相关性
主要内容
数据集:
Human Genome Diversity Project (HGDP) SNP 数据集
1064个个体
52个族群
原始数据:
1043个个体
每个个体 — 660,918 SNPs(163来自线粒体DNA,排除)— 用660,755
每个SNP — 2个等位基因 — 编码表示为:
目标一:
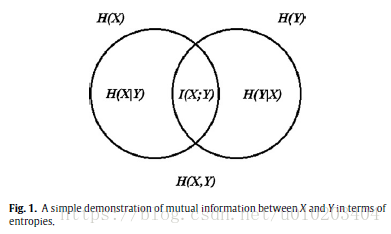
高分类准确度 — mutual information MI 互信息
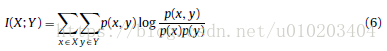
— 随机变量的熵
目标二:
基因组地理相关性 — principal components analysis PCA
由于维度较高 — 对PCA使用了“维度戏法”
—
维协方差矩阵
—
为中心数据矩阵,
— 特征向量
两边同乘
— 协方差矩阵
的第
个特征向量
两边同乘
可得: