ITK图像对齐
1、使用SimpleITK对齐图像
在看voxelmorph的代码,看到图像对齐部分,记录一下。
下面是从voxelmorph项目中截取的一段保存图像的函数。
函数输入分别是:配准后的图像、固定图像、要将配准图像保存的名字。
将图像对齐的操作需要将对齐的图像的原点、方向、间距设置成与 被对齐的图像一致。
def save_image(img, ref_img, name):
img = sitk.GetImageFromArray(img[0, 0, ...].cpu().detach().numpy())
img.SetOrigin(ref_img.GetOrigin())
img.SetDirection(ref_img.GetDirection())
img.SetSpacing(ref_img.GetSpacing())
sitk.WriteImage(img, os.path.join(args.result_dir, name))
这里只是做了属性的修改,并没有真正的对齐。
2、重采样并对齐图像
def align_seg_with_raw_nrrd(dcm, seg):
# Just for labelmap .... because of nearestNeighour interpolator
resampler = sitk.ResampleImageFilter()
resampler.SetReferenceImage(dcm)
resampler.SetTransform(sitk.Transform(3, sitk.sitkIdentity))
resampler.SetInterpolator(sitk.sitkNearestNeighbor)
seg_new = resampler.Execute(seg)
return seg_new
if __name__ == '__main__':
raw_dcm_file = r"path to your nrrd raw data"
seg_dcm_file= r"path to your seg file"
dcm = sitk.ReadImage(raw_dcm_file)
seg = sitk.ReadImage(seg_dcm_file)
print(raw_dcm_file, seg_new_file)
seg_new = align_seg_with_raw_nrrd(dcm, seg)
我要实现的目的是:将原始stl切下与原始数据重合的部分,并且用一个与原始数据一样的尺寸存放,并且对应的空间坐标一致(原点、spacing、direction)。
如下输入原始数据:

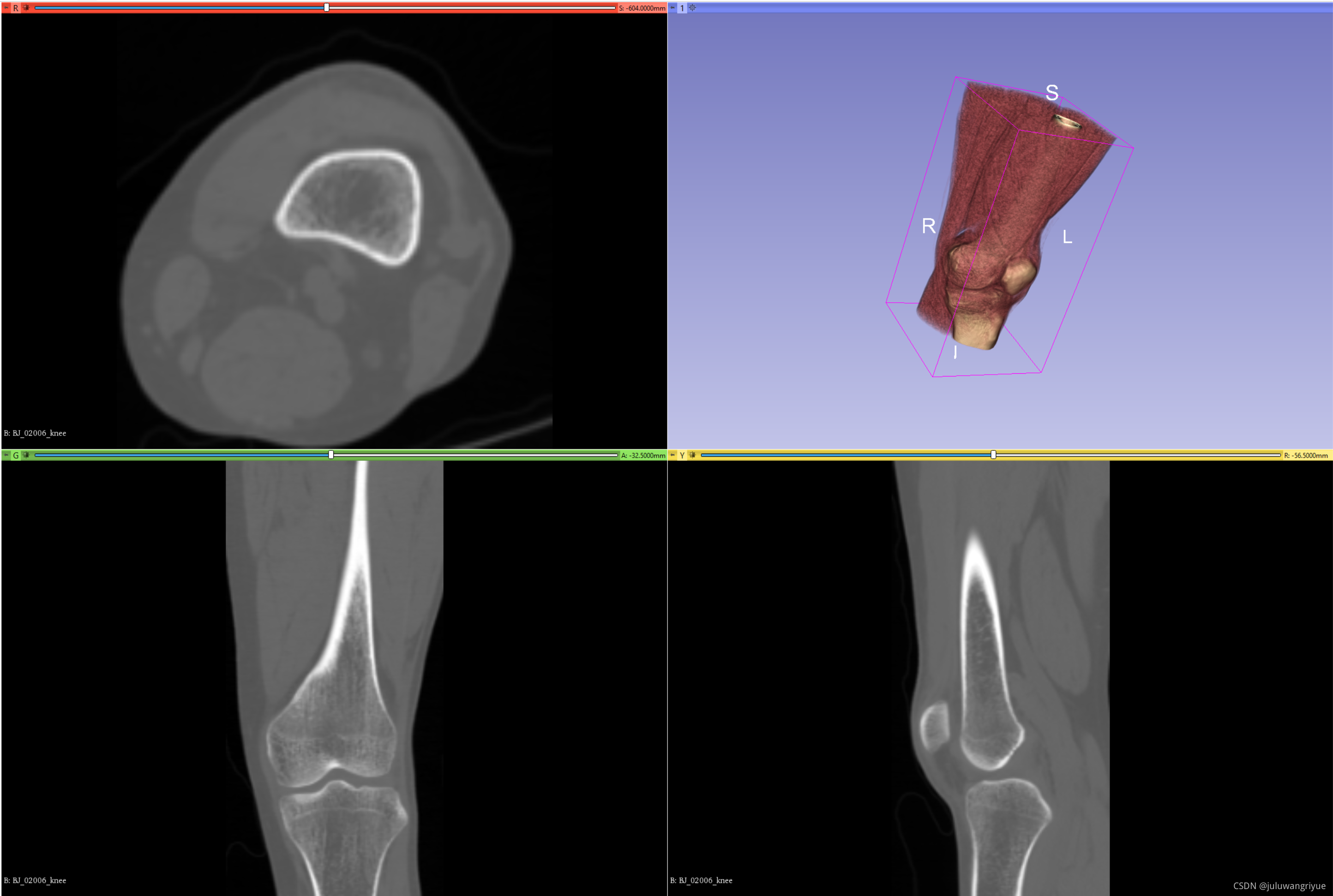
原始的stl数据,绿色部分:


对齐后的效果如下:
stl对齐后的volume信息如下

对齐后的渲染效果如下

3、中间走过的弯路
多设置参数,得到错误的结果。