作者:顾一文 审核:Listenlii
注:自iDIRECT方法的文章在今年出现以来,已经有若干公众号进行了解读。但全都集中于结果,而对我最感兴趣的方法部分都不涉及。本文主要从方法部分进行介绍。

Journal: PNAS
Published: January 6, 2022
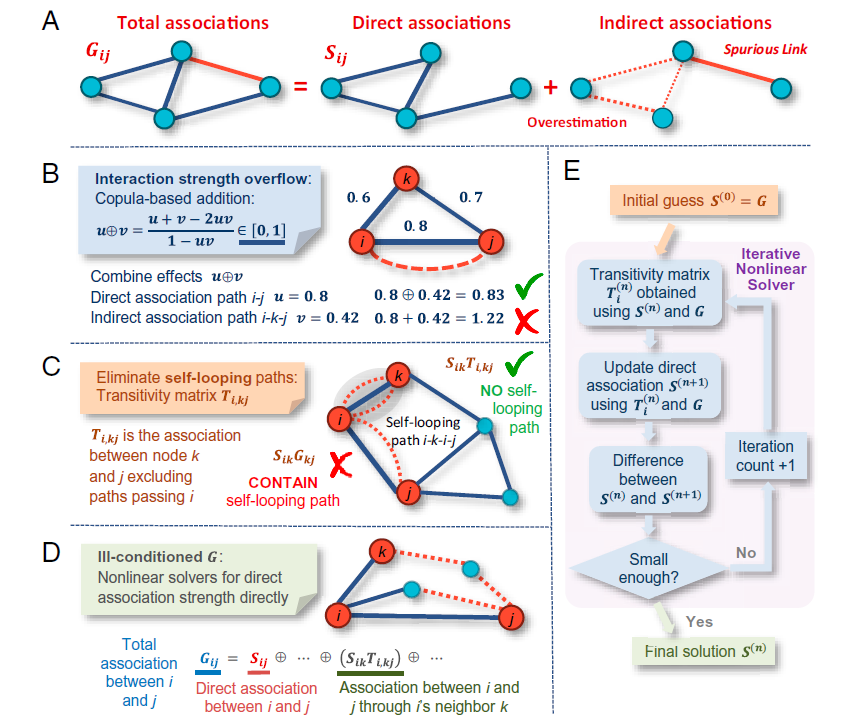
网络推理的目标是识别直接的联系及其优势,同时抑制间接的或传递的联系。
1921年,遗传学家、网络推理领域的创始人Sewall Wright描述了推断网络中错误链接的问题,他说:“两个变量之间的相关程度可以通过众所周知的方法计算,但是它仅给出了所有连接影响路径的结果”。例如,假设一个基因直接控制第二个基因,而第二个基因又直接控制第三个基因。相关性分析会错误地指出第一个基因直接影响第三个基因。通过衡量变量之间关系的方法,如皮尔森相关(Pearson correlation)、互信息(mutual information)、距离相关(distance correlation),我们会得到一个总相关依赖性矩阵G。但是这些方法没有办法直接区分其中的直接和间接相互依赖关系。
针对不能量化的问题,人们已经开发了一些方法来推断被测变量之间的直接关联,如偏相关(partial correlation),格兰杰因果(Granger causality),条件互信息(conditional mutual information CMI),部分互信息(part mutual information PMI),贝叶斯网络(Bayesian networks)。但是,这些方法的结果会因不同的数据集而有很大差异。此外,这些方法局限于研究局部依赖模式以识别潜在的间接边缘,并且只能考虑有限长度通常为2的间接路径。
为了解决有效性和普遍性的问题,又开发了几种使用关联矩阵的逆的更通用的方法来更好地估计直接依赖关系,包括(1)网络反卷积Network Deconvolution (ND),(2)全局沉默Global Silencing(GS), (3)生态关联推理的稀疏逆协方差估计SPIEC-EASI。从理论上讲,ND、GS和SPIEC-EASI提供了更通用的框架来估计观察到的总测量的直接影响,因此它应该更适用于各种应用中的网络推理。这三种方法具有明显的优势:首先,概念上,ND将间接影响视为沿着真实网络边缘的直接影响流,并将它们表示为直接相关矩阵的无限幂级数的总和,GS将测量的相关性视为小扰动并推导出类似于Modular Response analysis (MRA)的公式,而SPIEC-EASI则使用邻域选择或稀疏逆协方差选择来估计交互网络。这三种方法都能够考虑任意长度的间接路径。
由于网络推理的不确定性,总关联矩阵G趋于单一性或病态性。当G的单一性或病态性在实现过程中成为问题时,其他方法使用通用数值分析技术来反转关联矩阵G。例如,ND使用比例因子和基于特征分解的伪逆;GS使用引导随机化修改G;SPIEC-EASI遵循使用G的稀疏性的优化方法。通常的操作方法是由一个给定的总相关矩阵G和一个直接相关矩阵S,然后需找一个公式去联系G和S。在ND方法中,间接影响对应于所有间接路径的长度,例如:G= I+ S+ S2+S3+ ….,即S= (G- I) G-1,然后应用特征分解得到G-1。在GS方法中,节点i和节点j之间的相互关系分为两部分:节点i和节点j的一个邻居节点k之间的关联,以及节点k和节点j之间的关联,即矩阵乘积SG的非对角项。使用一些近似值,S根据G给出:S= (G + diag{G (G - I)}) G-1。在SPIEC-EASI方法中,S假定为G-1,然后G-1使用带有惩罚项的最小化过程来求解,假设G-1是稀疏的。这些方程中的差异主要是由于G中是否包括对角线项造成的。但是由于一些与ill-conditioning, self-looping, 和interaction strength overflow相关的问题,它们对间接关系的估计并不准确。
前人研究使用的三种方法的关键问题:

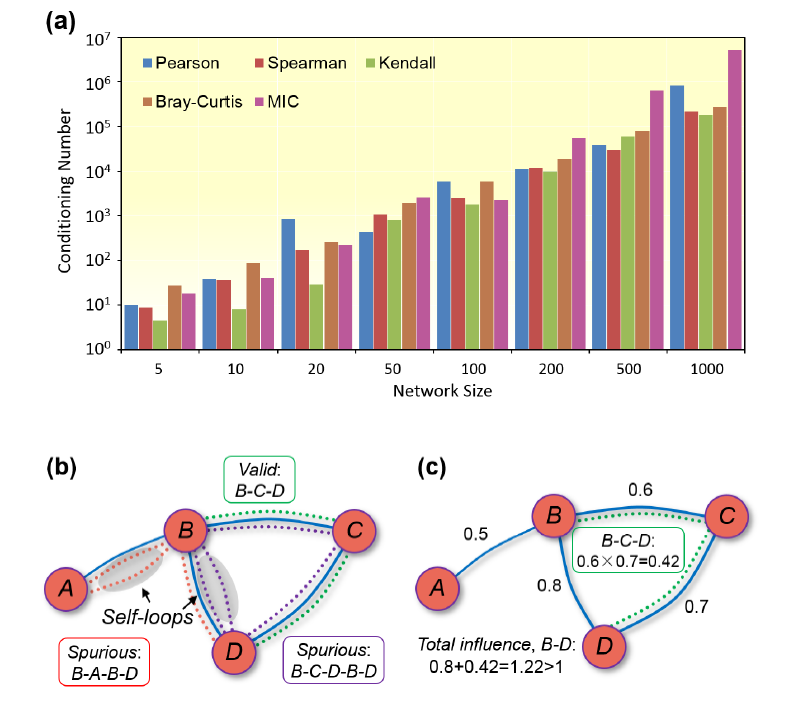
单一性(Singularity)指的是G的逆矩阵G-1, 使得GG -1 = G-1G = I的矩阵不存在; ill-conditioning指的是G的逆G-1是高度不可信的。一个矩阵的单一性可以通过检查它的秩是否小于它的大小,或者通过它的特征值是否包含0来检测。
1. 病态性的(ill-conditioning):因为总相关矩阵G是单一或病态的,所以ND/GS/SPIEC-EASI这些使用G-1矩阵的解决方法都是不可信的。
2. 自环性(self-loop):这些方法的计算公式都不能剔除包含自环连接的虚假的间接联系,这导致结果高估了间接关系的影响。
3. 相互作用强度溢出(interaction strength overflow):得到的直接关联矩阵S的相互关系数据理论上应该始终属于自然范围[0,1],但这些方法的结果实际上会超出这个范围。
Indirect如何解决上述三个问题并鉴别直接和间接关系
(a)当有了总相关矩阵G之后,把整个系统分成更小的子系统以便最小化不确定性因素的影响。因此不需要求总关联矩阵G的逆,避免了病态问题。并且通过两种非线性求解方法:T-solver,使用(B12)公式去解决当G给定时的传递性矩阵Ti; S-solver,使用(B14)公式解决当G和Ti给定时的直接相关矩阵S。

(b)此外通过传递性矩阵(Ti)去消除自循环诱发的间接关系。通过考虑两个节点i和j之间通过节点i的一个邻居k。节点i通过节点k和j之间的相互间接关系强度为SikTi,kj。其中Sik是i和k之间的直接关联强度,Ti,kj为节点k和j之间的关联强度,不包括经过i的路径。 SikTi,kj,不包括任何自循环诱导的间接路径,因为它们明确地排除在Ti,kj,之外。节点i和j之间的总相关强度Gij为节点i和j之间直接相关强度Sij加上通过节点k的间接相关Ti,kj。
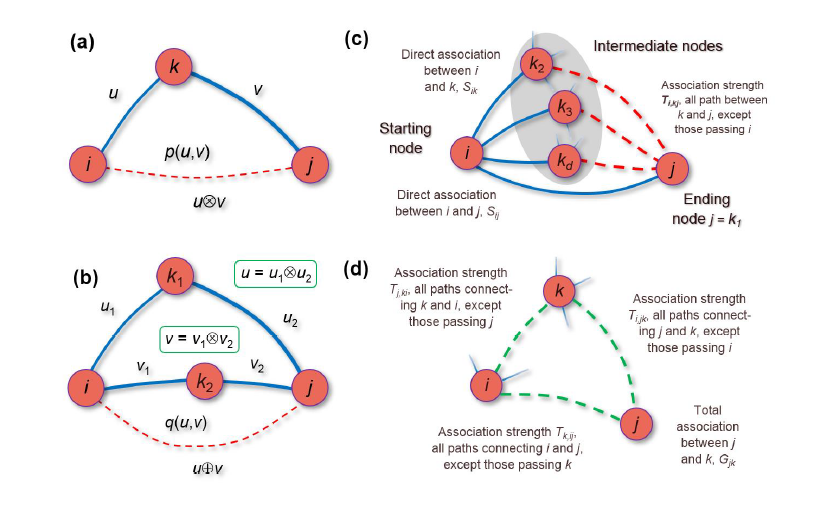
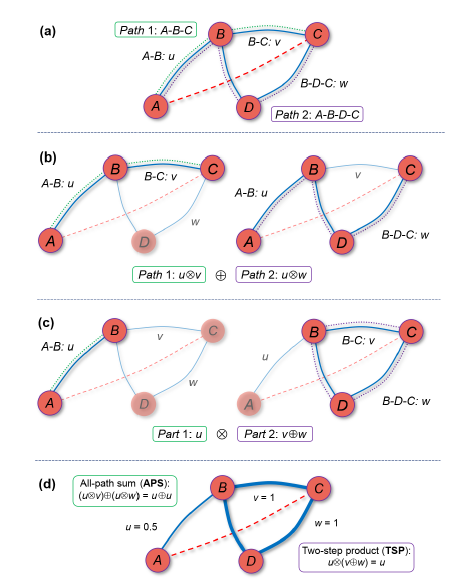
引入了两个新概念--顺序路径和平行路径。顺序路径:节点i和节点j通过中间节点k间接连接。i和j之间的间接关联强度为uUv。平行路径:节点i和j通过两条独特的路径间接连接在一起,这两条路径经过节点k1或节点k2。这两条路径的联合强度为u⊕v,其中u和v为每条路径的关联强度。节点k和节点j的总连接Gkj是Ti,kj加上Tj,kiTk,ij。由于Ti,kj定义中排除了节点i,因此消除了自环路引起的伪路径。
(c)解决相关强度溢出的问题。通过引入新的计算方法⊕和U去消除强度移除问题。
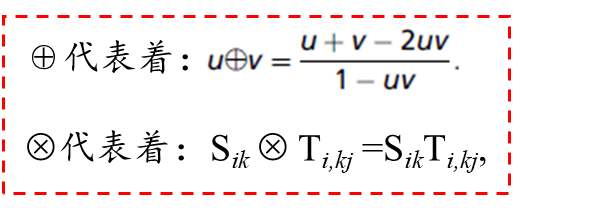
例如在下图B中,节点i和节点j之间的总相关为0.8⊕0.42=0.83,而以往的计算方法为:0.8+0.42>1,会造成相关程度溢出的问题。
且在计算节点i和节点j之间的总相关时,使用了两种策略,第一个策略:APS(the All-path Sum)策略,A和C总的相关强度为路径1和路径2的总和(u U v) ⊕(u U w);第二个策略:TSP(The Two-Step Product)策略,A和C总的相关强度为路径1和第二部分总路径和的乘积uU(v⊕w)。
总结性而言,iDIRECT是在给定相关家族很之后构建网络的过程中,通过筛选出直接和间接关系,使得我们得到的网络结构更加的趋于真实,节点之间的连线更加能代表生态关系。我们期望iDIRECT在网络科学、系统生物学和微生物学研究中有广泛的应用。
Ps: 目前该方法已经加入在MENA网络分析平台,且分析代码已经放在github上
网络分析方法最终的目的是希望创建一种方法,它可以在任意的不仅仅是线性模型中计算n个变量之间的依赖性关系,并且能够从复杂的关系中准确的量化直接相关和间接相关,从而能够准确的反应节点(OTU)在自然生态系统下的真实关系以及强度。

点分享

点点赞

点在看


猜你喜欢
10000+:菌群分析 宝宝与猫狗 梅毒狂想曲 提DNA发Nature Cell专刊 肠道指挥大脑
文献阅读 热心肠 SemanticScholar Geenmedical
16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流、快速解决科研困难,我们建立了“宏基因组”专业讨论群,目前己有国内外5000+ 一线科研人员加入。参与讨论,获得专业解答,欢迎分享此文至朋友圈,并扫码加主编好友带你入群,务必备注“姓名-单位-研究方向-职称/年级”。PI请明示身份,另有海内外微生物相关PI群供大佬合作交流。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍未解决群内讨论,问题不私聊,帮助同行。

学习16S扩增子、宏基因组科研思路和分析实战,关注“宏基因组”
点击阅读原文,跳转最新文章目录阅读