大家好,我是邓飞,这里介绍一下如何使用Haploview进行单倍型的分析。
计划分为三篇文章:
- 第一篇:Haploview做单倍型教程1–软件安装
- 第二篇:Haploview做单倍型教程2–分析教程
- 第三篇:Haploview做单倍型教程3–结果解读
今天是第二篇。
1. 数据准备
需要做单倍型分析的是基因型数据,一般是显著性的SNP,提取上下游500kb,然后进行block的分析。
这里,准备的是plink数据,比如我们要提取:
- 染色体是6
- 开始位置是1000000
- 终止位置是2000000
vcftools --vcf aaa.vcf --chr 6 --from-bp 1000000--to-bp 2000000--recode --out block1
将其转化为plink的map和ped数据:
plink --vcf block1.recode.vcf --recode --out a1
2. 整理数据
将map的第二列和第四列提取出来,保存为a1.info文件。
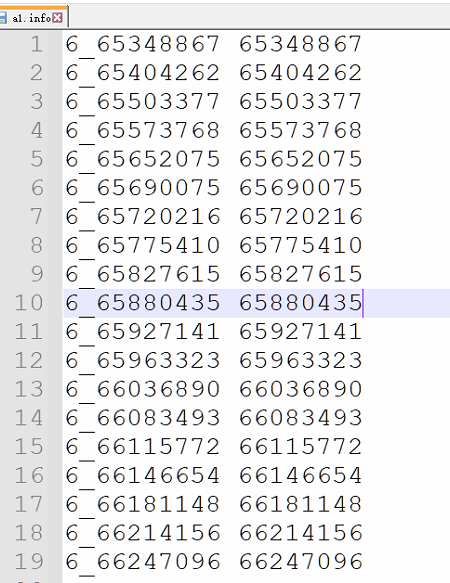
ped数据,保持不变:

3. 导入数据
选择第一种格式:Linkage Format,然后将ped数据导入到Data File中,将info数据导入到Locus Information File文件中。
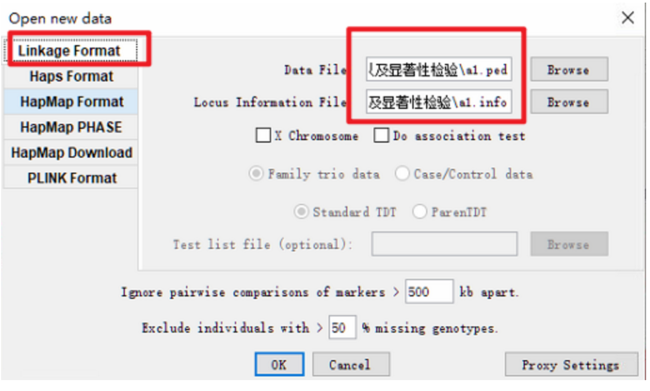
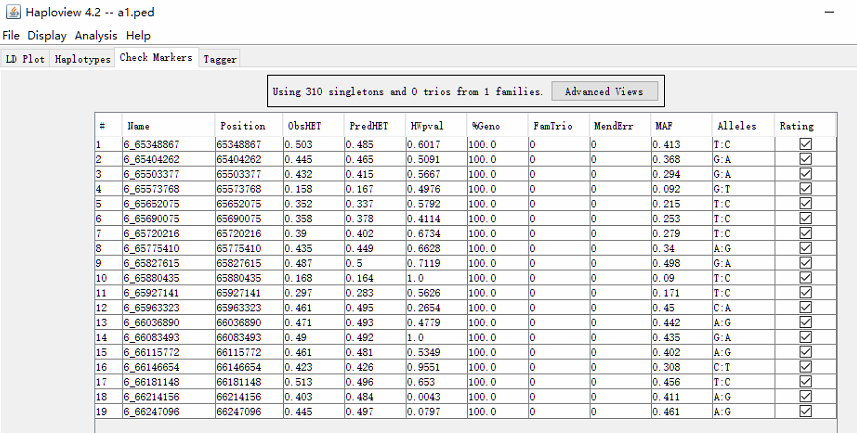
结果:

查看Block:

查看TaggerSNP:

上面就是下数据分析实操方法。
下一篇介绍Haploview结果如何解读及如何微调结果。
欢迎继续关注。