大家好,我是微学AI,今天给大家介绍一下深度学习实战39-U-Net模型在医学影像识别分割上的应用技巧,以细胞核分割任务为例。本文将介绍在医学影像分割领域中应用U-Net模型的方法。我们将从U-Net模型原理出发,并使用PyTorch搭建模型,详细展示模型代码。接着,我们将展示一些医学影像数据样例,并将它们加载到模型中进行训练。最后,我们将打印损失值与准确率,并对训练完的模型进行测试。
目录
- U-Net模型原理
- 使用PyTorch搭建U-Net模型
- 医学影像数据样例
- 数据加载与预处理
- 模型训练
- 模型测试
- 总结
1. U-Net模型原理
U-Net是一种用于图像分割的卷积神经网络(CNN),U-Net在医学图像分割领域具有广泛的应用。其网络结构包括一个收缩路径(编码器)和一个扩张路径(解码器)。
编码器负责提取图像中的特征,由多个卷积层、ReLU激活函数和最大池化层组成。而解码器负责将提取到的特征映射回原始图像的空间尺寸,由多个卷积层、ReLU激活函数和上采样层组成。此外,U-Net中还有跳跃连接,将编码器和解码器之间的特征连接起来,以帮助提高分割精度。
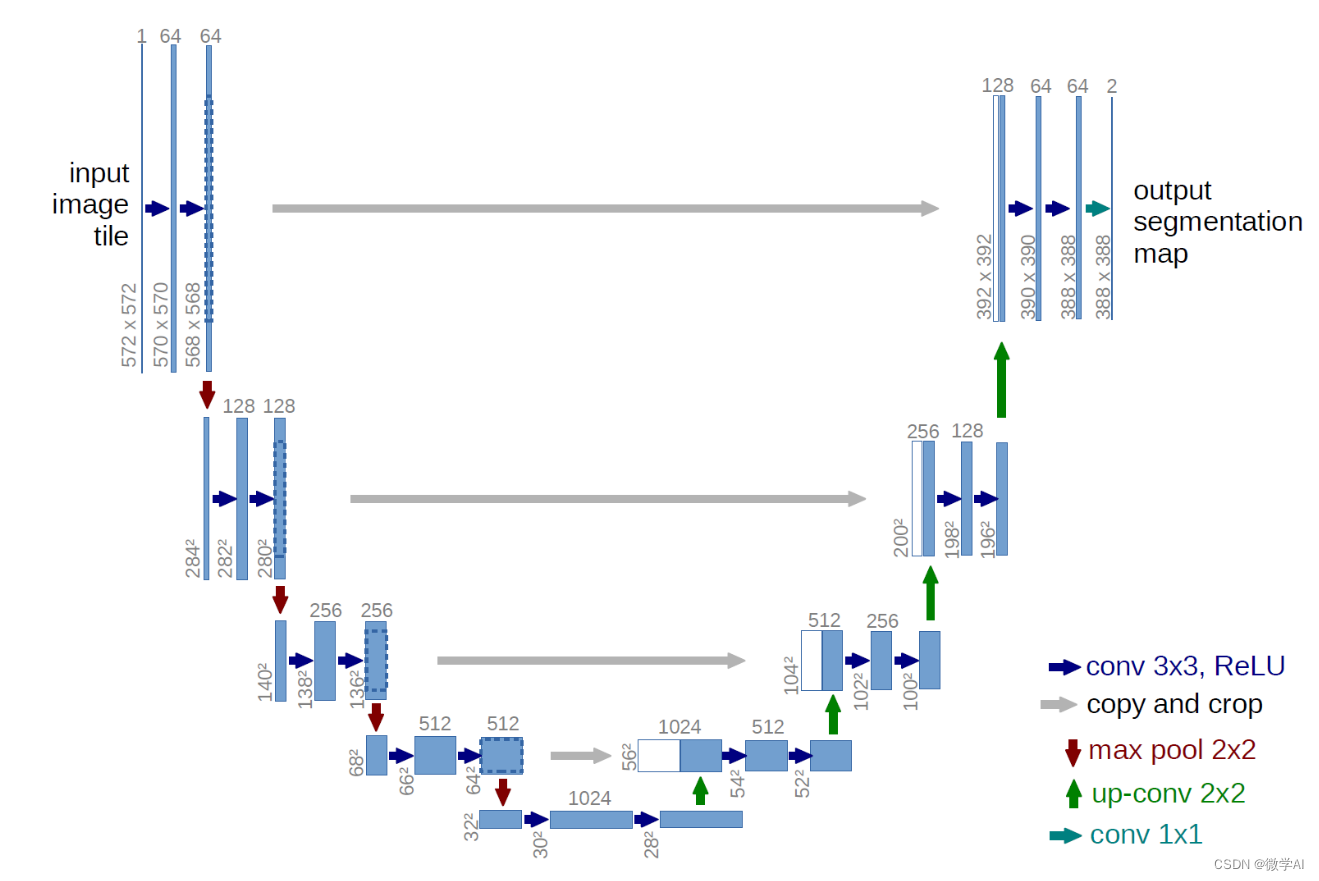
U-Net模型的数学原理可以用以下公式表示:
首先,U-Net模型由一个编码器和一个解码器组成。编码器部分负责将输入图像逐渐降采样为较小的特征图,而解码器部分则将这些特征图逐渐