1.关于因变量y的异常点
我们可以直接用R软件的rstudent()函数计算出删除学生化残差 的数值, 的观测值即判定为异常值。
代码实现如下:
data2.2<-read.csv("C:/Users/Administrator/Desktop/data2.2.csv",head=TRUE)
lm2.2<-lm(y~x,data=data2.2)
rstudent(lm2.2)
输出结果为:
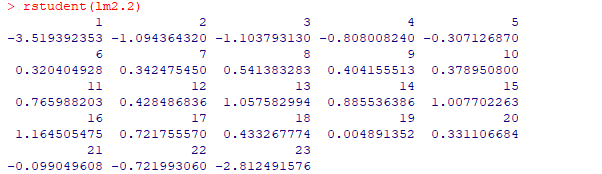
从输出结果可以看出,1号数据应该判定为异常点,直接删除。
2.关于自变量x的异常值
我们引入Cook距离,用来判断强影响点是否为
的异常值点。Cook距离的计算公式为:
对于Cook距离大小标准的判定比较复杂,一个粗略的标准是:当
时,认为不是异常点;反之则认为是异常点。
实现代码如下:
data2.2<-read.csv("C:/Users/Administrator/Desktop/data2.2.csv",head=TRUE)
lm2.2<-lm(y~x,data=data2.2)
hii<-hatvalues(lm2.2) # 计算杠杆值
cooks.distance(lm2.2) # 计算Cook距离
输出结果如下:
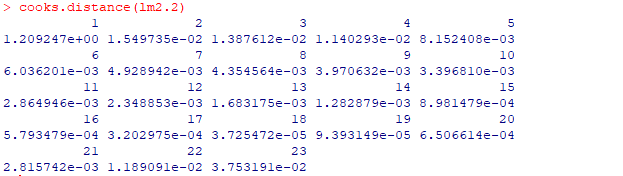
从中可以看出,1号数据的Cook距离是大于0.5的,所以该点应该直接删除。