Entrez是一个搜索引擎,国家生物技术信息中心(NCBI)网站集成了 几个健康科学的数据库,如:如“科学文献,DNA和蛋白质序列数据库, 蛋白质三维结构,蛋白质结构域的数据,表达数据,基因组完整拼接本等。
Entrez的编程工具”(eUtils):通过它把搜索的结果返回到自己编写的程序里面, 需要提供URL,并且自己解析XML文件。 Entrez模块,利用该模块可以省去提供URL和解析XML的步骤。
Entrez模块中的函数, 同时也是eUtils中具有的一些函数:
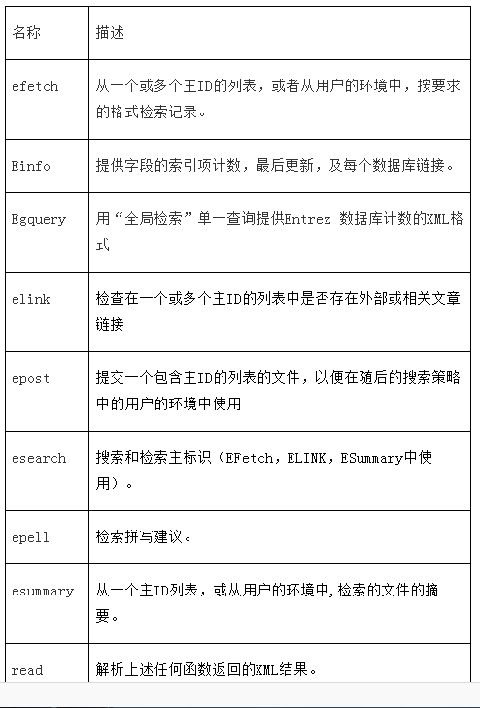
从pubmed中查找相关文献, 所有返回的结果用Entrez.read()解析
from Bio import Entrez
my_em = '[email protected]' db = "pubmed"
# Search Entrez website using esearch from eUtils
# esearch returns a handle (called h_search) 主要用来返回id,
h_search = Entrez.esearch(db=db, email=my_em,
term="python and bioinformatics")
record = Entrez.read(h_search) # Parse the result with Entrez.read(),record是字典
res_ids = record[“IdList”] # Get the list of Ids returned by previous search. 该键的值是列表
# For each id in the list
for r_id in res_ids:
# Get summary information for each id
h_summ = Entrez.esummary(db=db, id=r_id, email=my_em)
# Parse the result with Entrez.read()
summ = Entrez.read(h_summ) #返回一个列表,第一个元素是字典,不同的数据库返回的数据的结构不一样
print(summ[0]['Title'])
print(summ[0]['DOI'])
print('==============================================')
结果:
do_x3dna: A tool to analyze structural fluctuations of dsDNA or dsRNA from molecular dynamics simulations. 10.1093/bioinformatics/btv190
==============================================
RiboTools: A Galaxy toolbox for qualitative ribosome profiling analysis.
10.1093/bioinformatics/btv174
==============================================
Identification of cell types from single-cell transcriptomes using a novel clustering method. 10.1093/bioinformatics/btv088
==============================================
Efficient visualization of high-throughput targeted proteomics experiments: TAPIR.
10.1093/bioinformatics/btv152