暂时总结线上竞赛部分,等面试完后再看情况是否更新这篇文章。
因为涉及隐私数据,所以我只能边提思路边提供自己的部分代码,数据方面就无能为力啦。
实际用到的代码进过了多重改动,所以本文的代码会出现部分命名混乱的情况,思路为主,不要在意这种小问题。
FinTech是什么
简单地说,它是招行为了选拔训练营的人才而设立的 竞赛 。这是我第一次参加实战,本来也没奢望能拿到面试资格,没想到再过几天就要去面试了,姑且总结下思路与经验,以备往后温习和各位借鉴。大佬们欢迎留言或私信我交流更好的思路呀~
线上竞赛思路
选题
- 第五题 识别财务造假
- 样本数量 7653,其中正负样本数量比约 1:70,只有少数缺失值
- 提供了34个特征,标签为 ”fake“,1 表示财务造假,0 表示没有造假
- 评分标准:
其中
表示的是全部实际为正的样本中,成功被预测(召回)的比例,如果有”漏网之鱼“的代价很大,那么通常要求 Recall 足够高!
表示的是预测为正的样本中,实际上确实为正样本的比例,如果”错杀“的代价很大,那么就要求 Precision足够高!
一番权衡下,就有了新的指标 。
尝试一
不做任何特征工程与数据预处理,连缺失值也不管,数据集也没划分,直接开干,轮番将 knn, svm, mlp, naive bayes, randomforest, adaboost, xgb 都用默认参数进行拟合预测,天,accuracy妥妥的 90%+,一提交,稳了,直接零分。。。。。。
用 看了下,只有 109 条正样本,明显的数据集不平衡。赶紧查了下数据集失衡下的 常规操作 ,首选的两个做法是重采样以及合理地进行特征选择。
这一部分的处理流程主要是:特征选择——标准化——离散化——欠采样——训练——过采样——训练。
先用直方图查看下数据分布,我随便挑选了其中四个有代表性的分布:
import matplotlib.pyplot as plt
cols = features.columns
for col in cols:
plt.hist(features[col], bins=200)
plt.show()




看起来每个特征的分布很离散,有很明显的离群值,我最初觉得这些离群值可能有重要的区分作用,所以没有将它去除(BTW:这些离群值也许真的是高分的关键——毕竟第一名 0.77 远远把第二名甩在后头……只是我还不知道怎么利用离群值来辅助分类,大佬们如果有好的想法赶紧留言或给我私信!!!!)。
但我还是将所有特征视为一个整体做了一次异常值检测,用的是 LOF,只检测出两个异常样本,直接删除。在这一步之前还有个缺失值处理的过程,只有一个特征的3个值缺失,我将其设为 0。
from sklearn.neighbors import LocalOutlierFactor as LOF
lof = LOF(n_neighbors=35, contamination=0.05, n_jobs=-1)
# y_pred = lof.fit_predict(np.reshape(features.A_to_L_ratio, (-1, 1)))
# for i in range(len(features.columns)):
copy_ = features.iloc[:, 0:1].copy()
y_pred = lof.fit_predict(copy_)
copy_.iloc[y_pred, 0:1] = np.nan
copy_data.dropna(how='any', inplace=True)
copy_data.reset_index(inplace=True)
copy_data.drop(columns=['index', 'stockcode'], inplace=True)
下一步,用 pandas.corr() 输出协方差矩阵,将与标签相关性最低的若干个特征 drop 掉。
more_related_cols = []
indices = correlation.index
for i in range(len(cols)):
if abs(correlation[i]) > 0.01:
more_related_cols.append(indices[i])
# 这里保留了 13 个特征
more_related_cols
进一步地,我还分别用了统计学的 f 值和信息论的互信息来进一步筛选,下面的代码是使用 f 值的部分,选出 10 个数值最高的特征。
from sklearn.feature_selection import mutual_info_classif, f_classif, SelectKBest
select = SelectKBest(f_classif, k=10)
select.fit(r_features, r_labels)
# select.scores_
more_related_cols_from_f_classif = [more_related_cols[i] for i in range(len(more_related_cols)) if i in [0, 1, 2, 4, 5, 6, 7, 8, 10, 11, 12]]
more_related_cols_from_f_classif
下一步是标准化。因为一时半会还没想明白哪种方法合适,索性全部用了 MinMax。
'''standardization'''
from sklearn.preprocessing import MinMaxScaler
mm = MinMaxScaler()
r2_features = mm.fit_transform(r_features[more_related_cols_from_f_classif])
r2_features.shape
这里保险起见,我对特征矩阵单独再用了 corr() 剔除了相关性在 0.75 以上的特征。
下一步是离散化。我才发现 sklearn 里面集成了等距、等频、聚类三大类,牛逼。因为数值总体非常离散,而有的又非常集中,感觉等距的话受离群值影响很大,等频的也不合适,所以我首选的是聚类法。
from sklearn.preprocessing import KBinsDiscretizer
kb = KBinsDiscretizer(n_bins=10, encode='ordinal', strategy='kmeans')
df_r2[more_related_cols_from_f_classif] = kb.fit_transform(df_r2[more_related_cols_from_f_classif])
现在终于要采样训练了。第一次用 imblearn,什么都不懂,用了最基础的随机欠采样方法,将正负样本比例控制在 0.2~0.9以内(都试试)。
from imblearn.under_sampling import RandomUnderSampler
resample = RandomUnderSampler(0.2)
r3_features, r3_labels = resample.fit_resample(df_r2[more_related_cols_from_f_classif], df_r2['fake'])
r3_features.shape, r3_labels.shape
为了知道它的泛化效果,我当时将样本划分出 30% 的验证集。注意!!!这个操作其实非常不合理!!!我是后边看了好几篇 kaggle 的文章才意识到的,数据集的划分应该是放在重采样之前,毕竟我们最终的目的是在官方给定的测试集上取得好的泛化效果,如果在已经重采样过的数据集上测试,其实样本的分布与官方测试集可能非常大,所以结果毫无意义。更合理的方式请参照尝试二与三。
from sklearn.model_selection import train_test_split
# 其实这里的 test 应该换成 val 比较合适
f_train, f_test, l_train, l_test = train_test_split(r3_features, r3_labels, test_size=0.3, random_state=42)
终于到了激动人心的训练环节。默认参数来一波:
from sklearn.ensemble import RandomForestClassifier
rf = RandomForestClassifier(n_estimators=200)
rf.fit(f_train, l_train)
print('Result from rf')
print(metrics.classification_report(l_train, rf.predict(f_train)))
print(metrics.classification_report(l_test, rf.predict(f_test)))
完整的结果太长了,我整理成了表格,每个空格中,划线左侧的数值为训练集上结果,右侧的为验证集上的结果:
| 模型 | rf | mlp | bernoulliNB | svc | xgb | ada |
|---|---|---|---|---|---|---|
| precision | 1.00/0.56 | 0.79/0.52 | 0.63/0.60 | 0.91/0.89 | 0.99/0.99 | 1.00/1.00 |
| recall | 1.00/0.50 | 0.78/0.60 | 0.35/0.45 | 1.00/1.00 | 0.97/0.97 | 1.00/0.99 |
| f1 | 1.00/0.53 | 0.78/0.56 | 0.45/0.51 | 0.95/0.94 | 0.98/0.98 | 1.00/1.00 |
似乎除了前两个以外,其他都没有过拟合。而且后边三个模型的结果简直不要太诱人好嘛!听说排行版上第一名才 0.77??!!我赶忙对测试集做了标准化和离散化的 transform 处理然后急不可耐地提交。However,三次提交的结果在 0.03~0.06之间。
WHY?验证集上的结果挺好的不是?问题就在于前面提到的,验证集也是重采样过的,其正负样本分布情况已经变形了,对是否过拟合的测试完全无意义。(话虽这么说,但当时并没有反应过来,以为是参数的锅,疯狂地调参调了一天……)
上面展示的是欠采样的结果,至少还能在 0.06 的分值,后边试了过采样直接就 0 分,过拟合更严重,但是思路错了导致分析不出问题在哪。
尝试二
找了几篇 kaggle 上处理金融诈骗的分享文章,同样是数据不平衡,但它们的数据集是百万量级的,而正样本只有几百,但最后的拟合结果同样好到飞起,所以我一度以为我能很快逆袭。 这都是后话了,放在反思部分再说。
这一次我换了一种数据观察的方法——画出正负两种样本的概率直方图,剔除没有区分度的特征(不过要注意别把标题看成横坐标了,我一开始就因此删错了特征)。下面这段代码基本上来自 kaggle 上某一篇大神的文章:
#Select only the anonymized features.
import matplotlib.pyplot as plt
import matplotlib.gridspec as gridspec
import seaborn as sns
cols = norm_data.drop(columns=['fake']).columns
plt.figure(figsize=(16, len(cols)*4))
gs = gridspec.GridSpec(len(cols), 1)
for i, cn in enumerate(norm_data[cols]):
# print(i, cn)
ax = plt.subplot(gs[i])
sns.distplot(norm_data[cn][norm_data["fake"] == 1], bins=50)
sns.distplot(norm_data[cn][norm_data["fake"] == 0], bins=100)
ax.set_xlabel('')
ax.set_title('histogram of feature: ' + str(cn))
plt.show()
摘几个图来感受下,有一些很正态,但也有的非常极端:
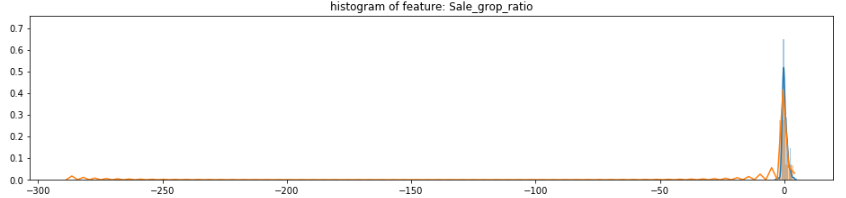
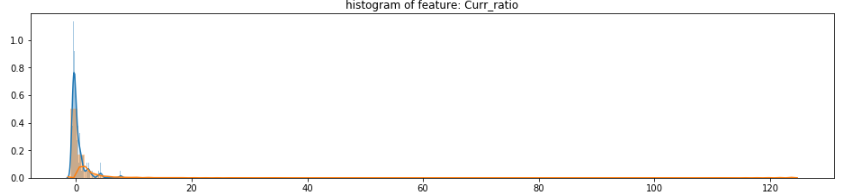

这一步大概剔除了 10 个特征。
在前面的尝试中,完全的欠采样会导致大量信息丢失,因而欠拟合;而过采样会导致正样本信息被不断重复,因而过拟合。所以在后边的比赛中,我尝试了先用欠采样将负样本稍微筛选一部分,再在此基础上用过采样。为了找到最佳的搭配,我用了两重 for 循环去迭代不同的采样比例。
方便起见,我将两个采样的过程定义为函数,不过我并没花时间了解不同采样技术的原理,所以可能没有选到恰当的方法。
过采样:
from imblearn.over_sampling import ADASYN, SMOTE, BorderlineSMOTE, SVMSMOTE, RandomOverSampler
def over_resample(f_train, l_train, ratio=0.5):
print('Original dataset shape: %s' % Counter(l_train))
over_resample = SMOTE(random_state=0, sampling_strategy=ratio)
r2_features, r2_labels = over_resample.fit_resample(f_train, l_train)
print('SMOTE Resampled dataset shape: %s' % Counter(r2_labels))
# over_resample = ADASYN(n_neighbors=5, n_jobs=-1, random_state=0, sampling_strategy=ratio)
# r2_features, r2_labels = over_resample.fit_resample(r_features, r_labels)
# print('ADASYN Resampled dataset shape: %s' % Counter(r2_labels))
# over_resample = BorderlineSMOTE(k_neighbors=2, n_jobs=-1, random_state=0, sampling_strategy=0.05)
# r2_features, r2_labels = over_resample.fit_resample(r_features, r_labels)
# print('BorderlineSMOTE Resampled dataset shape: %s' % Counter(r2_labels))
# over_resample = SVMSMOTE(k_neighbors=2, n_jobs=-1, random_state=0, sampling_strategy=0.04)
# r2_features, r2_labels = over_resample.fit_resample(r_features, r_labels)
# print('SVMSMOTE Resampled dataset shape: %s' % Counter(r2_labels))
# over_resample = RandomOverSampler(n_neighbors=20, n_jobs=-1, random_state=0, sampling_strategy=0.1)
# r2_features, r2_labels = over_resample.fit_resample(r2_features, r2_labels)
# print('RandomOverSampler Resampled dataset shape: %s' % Counter(r2_labels))
return r2_features, r2_labels
欠采样:
'''resample on train set'''
from imblearn.under_sampling import RandomUnderSampler, InstanceHardnessThreshold, NearMiss, NeighbourhoodCleaningRule, OneSidedSelection, TomekLinks
from collections import Counter
from sklearn.tree import DecisionTreeClassifier
from sklearn.linear_model import LogisticRegression
def under_resample(f_train, l_train, ratio=0.5):
print('Original dataset shape: %s' % Counter(l_train))
under_resample = TomekLinks(n_jobs=-1, random_state=0)
r_features, r_labels = under_resample.fit_resample(f_train, l_train)
print('TomekLinks Resampled dataset shape: %s' % Counter(r_labels))
# under_resample = InstanceHardnessThreshold(n_jobs=-1, random_state=0)
# r_features, r_labels = under_resample.fit_resample(r_features, r_labels)
# print('InstanceHardnessThreshold Resampled dataset shape: %s' % Counter(r_labels))
# under_resample = NeighbourhoodCleaningRule(n_jobs=-1, random_state=0, n_neighbors=50)
# r_features, r_labels = under_resample.fit_resample(r_features, r_labels)
# print('NeighbourhoodCleaningRule Resampled dataset shape: %s' % Counter(r_labels))
# under_resample = OneSidedSelection(n_jobs=-1, random_state=0, n_neighbors=1)
# # r_features, r_labels = under_resample.fit_resample(r_x, df_r2['fake'])
# r_features, r_labels = under_resample.fit_resample(r_features, r_labels)
# print('OneSidedSelection Resampled dataset shape: %s' % Counter(r_labels))
under_resample = RandomUnderSampler(random_state=0, sampling_strategy=ratio)
r_features, r_labels = under_resample.fit_resample(r_features, r_labels)
print('RandomUnderSampler Resampled dataset shape: %s' % Counter(r_labels))
return r_features, r_labels
正如前面所说的,在采样前就要划分数据集,留出一部分【原生态】的样本作为验证集。我当时没有想得很明白验证集与测试集的区别,所以将原始数据划分为了训练集、验证集与测试集。但后边意识到测试集其实就是官方的测试样本,所以这里的【验证集与测试集】我在后边就合起来作为一个验证集了(估计没有比我骚的操作了……)
'''Re-splitting raw data into training, validating, testing set'''
from sklearn.model_selection import train_test_split
f_train, f_test, l_train, l_test = train_test_split(dropped_copy_data_2.drop(columns=['fake']), copy_data.fake, test_size=0.3, random_state=0)
f_test, f_val, l_test, l_val = train_test_split(f_test, l_test, test_size=0.33, random_state=42)
与这一步相对应的,需要自定义一个验证方式用于 GridSearchCV 等步骤时作为 cv 参数的传入值:
'''stack resampled set and test set; predefine cv'''
from sklearn.model_selection import PredefinedSplit
def my_cv(r_features, f_test, r_labels, l_test):
train_val_features = np.concatenate((r_features, f_test), axis=0)
train_val_labels = np.concatenate((r_labels, l_test), axis=0)
# 0 indicates validation set. Data with indeices -1 wont be split into validation set
test_fold = np.zeros(train_val_features.shape[0])
test_fold[:r_features.shape[0]] = -1
pfs = PredefinedSplit(test_fold=test_fold)
return train_val_features, train_val_labels, pfs
这里要强调另一个顺序相反的:corr() 的使用。这个操作反而建议在采样、并且标准化过后再进行,因为在极度不平衡、而且不同特征的数量级相差较大的数据集中,相关性的计算过程会以样本多的主导。
对上面经过直方图观察筛选后的数据进行标准化,这一次我挑选出了一部分用 MinMax,一部分用 Standard,剩下的不标准化。
norm_data = copy_data.copy()
'''standardization'''
from sklearn.preprocessing import MinMaxScaler, StandardScaler
std = StandardScaler()
mm = MinMaxScaler()
norm_data[mm_cols] = mm.fit_transform(norm_data[mm_cols])
norm_data[std_cols] = std.fit_transform(norm_data[std_cols])
norm_data.describe()
这个时候再用 corr() 结果就合理多了。
(以上是特征选择的过程,在比赛中我还试过了 RFECV 配合 AdaBoost 进行递归特征消除,然而结果不怎样;另外还试了降维的方法:PCA,LDA,还专门降到二维来可视化看这两种方法有没有明显的区分性。但是因为其他步骤的处理不尽合理,所以我当时没有看出这些方法有带来什么作用。)
特征选择完成后,依次是数据集划分——标准化(与上面的标准化不同,上面的标准化是为了进行特征选择,是对全部样本进行;下面的标准化是对训练集用 fit_transform,对验证集用 transform)——离散化——欠采样 & 过采样——训练。
第一次尝试的采样组合为:
'''Re-undersampling and predefine indices for cv'''
r_features, r_labels = under_resample(f_train, l_train, 0.04)
r2_features, r2_labels = over_resample(r_features, r_labels, 0.6)
f_train_val, l_train_val, pfs = my_cv(r2_features, f_val, r2_labels, l_val)
新的标准化代码为:
'''standardization'''
from sklearn.preprocessing import MinMaxScaler, StandardScaler
std = StandardScaler()
mm = MinMaxScaler()
f_train_1 = mm.fit_transform(f_train[mm_cols])
f_test_1 = mm.transform(f_test[mm_cols])
f_val_1 = mm.transform(f_val[mm_cols])
f_train_2 = std.fit_transform(f_train[std_cols])
f_test_2 = std.transform(f_test[std_cols])
f_val_2 = std.transform(f_val[std_cols])
f_train_3 = f_train[['Pre_np', 'Opinion']].values
f_test_3 = f_test[['Pre_np', 'Opinion']].values
f_val_3 = f_val[['Pre_np', 'Opinion']].values
print(f_train_1.shape, f_train_2.shape, f_train_3.shape)
f_train = np.concatenate((f_train_1, f_train_2, np.reshape(f_train_3, (-1, 2))), axis=1)
f_test = np.concatenate((f_test_1, f_test_2, f_test_3.reshape(-1, 2)), axis=1)
f_val = np.concatenate((f_val_1, f_val_2, f_val_3.reshape(-1, 2)), axis=1)
f_train.shape
训练的过程依旧是先用默认参数,不过这次我用了迭代的方式:
method = {'knn': KNeighborsClassifier(), 'mlp': MLPClassifier(), 'bnb': BernoulliNB(), 'svm': SVC()}
for k, v in method.items():
clf(v, r2_features, f_test, r2_labels, l_test)
fitting(v, np.concatenate((f_test, f_val)), np.concatenate((l_test, l_val)))
其中 clf 和 fitting 是新定义的函数:
# from sklearn.ensemble import VotingClassifier
from sklearn import metrics
def clf(estimator, f_train, f_test, l_train, l_test):
estimator.fit(f_train, l_train)
print('Result from %s' % estimator)
print(metrics.classification_report(l_train, estimator.predict(f_train)))
print(metrics.classification_report(l_test, estimator.predict(f_test)))
def fitting(estimator, f_test, l_test):
print('Result from %s' % estimator)
print(metrics.classification_report(l_test, estimator.predict(f_test)))
这里的训练结果分数明显就低得多了(合理多了),挑选出在验证集上分值最高的 svc 与 knn进行参数遍历:
'''potential better models: knn, svc'''
params = {'C': np.logspace(-2, 4, 7),
'gamma': np.logspace(-10, -1, 9),
'class_weight': [{0:1, 1:1}, {0:1, 1:1.25}, {0:1, 1:1.5}, {0:1, 1:1.75}, {0:1, 1:2}],
'max_iter': [50, 100, 150, 200]
}
best_params_svc = grid_search(SVC(random_state=42), f_train_val, l_train_val, params, cv=pfs, scoring='f1')
svc = SVC(random_state=42, **best_params_SVC)
clf(svc, r2_features, f_test, r2_labels, l_test)
fitting(svc, np.concatenate((f_test, f_val)), np.concatenate((l_test, l_val)))
其中的 grid_search 为自定义的函数:
from sklearn.model_selection import GridSearchCV
def grid_search(estimator, features, labels, params, cv=5, scoring='f1'):
cv = GridSearchCV(estimator, params, cv=cv, scoring=scoring, n_jobs=-1)
cv.fit(features, labels)
print(cv.best_params_)
print(cv.best_estimator_)
print(cv.best_score_)
return cv.best_params_
训练结果不太满意,对采样率作了调整,具体为通过迭代的方式:
under = np.linspace(0.02, 0.05, 10)
over = np.linspace(0.5, 0.9, 5)
for i in under:
for j in over:
print('Under ratio %.3f, over ratio %.2f' % (i, j))
r_features, r_labels = under_resample(f_train, l_train, i)
r2_features, r2_labels = over_resample(r_features, r_labels, j)
# f_train_val, l_train_val, pfs = my_cv(r2_features, f_val, r2_labels, l_val)
f_train_val, l_train_val, pfs = my_cv(r2_features, np.concatenate((f_test, f_val)), r2_labels, np.concatenate((l_test, l_val)))
params = {'criterion': ['gini', 'entropy'],
'max_depth': [1, 2],
'min_samples_split': [20, 30, 40],
'min_samples_leaf': [15, 20, 30],
'class_weight': [{0:1, 1:1}, {0:1, 1:2}, {0:1, 1:2}],
}
best_params_rf = grid_search(RandomForestClassifier(n_estimators=20, n_jobs=-1, random_state=42), f_train_val, l_train_val, params, cv=pfs, scoring='f1')
print('\n')
然而又是一番骚操作过后,结果并没有想象中的好,提交的结果大概为 0.05~0.06,期间有一次用 MLP 胡乱搞的并且一时没注意不小心就提交了,结果反而达到了0.09,实在是让人不得不相信玄学……
尝试三
如果你从前面一直看下来,你会发现明显少了一个重要步骤:离群值检测。
我前面一直没有做这一步,是因为我一直迷之坚信这些离群值会对正负样本的区分起到关键作用。虽然不确定到底是我猜错了还是我没有找到正确利用离群值的打开方式(后者更有可能),在比赛最后两天,心如死灰的我还是决定试试做这一步。
去除离群值的方法主要可以有基于 LOF,Isolation Forest,One-Class SVM,Z-Score,DBSCAN,Box 等。我采用的两种是基于 Z-Score 与 Box。
- 基于 Z-Score 是指默认数据分布符合正态分布,将某个分位点以外的数值当作离群值。0.05 分位点对应的横坐标为 1.95,从一篇文章中学到通常将阈值设置为 2.5/3.0/3.5。
- 基于 Box 是指将每个特征画成箱形图,将 区间外的数值当作离群值,通常取 c 为 1.5,我分别遍历了 {1.5, 3.0, 4.5} 等取值。
而对于离群值的处理,一开始我是直接 drop 掉,结果在验证集上非常漂亮,大概在 0.20~0.40 的范围。但是后边意识到 drop 掉的太多了,将近一半,可能会过拟合,所以又改为填充法(均值、众数和贝叶斯随机填充)。现在想想挺后悔没有试试用 drop 的方法训练到的模型去提交一次的,说不定逆袭就在一举(抱歉我膨胀了)。
结合了离群值检测的完整代码为:
'''Z-score based'''
def thrunk_z_score(data, thre):
data[(data<-thre) | (data>thre)] = np.nan
'''Box based'''
def thrunk_box(data, thre):
df_data = pd.DataFrame(data)
box = df_data.boxplot(return_type='dict')
col = df_data.columns
# len(box['fliers']) = 属性个数
for i in range(len(box['fliers'])):
# boxplot 中把截断点到分位点之间的连线形象地称为胡须(即 whiskers),box['whiskers'] 总共有(2 * 属性个数)个 values
lower_bound = box['whiskers'][i * 2].get_ydata()[1]
lower_bound -= (box['whiskers'][i * 2].get_ydata()[0] - box['whiskers'][i * 2].get_ydata()[1]) * thre
upper_bound = box['whiskers'][i * 2 + 1].get_ydata()[1]
upper_bound += (box['whiskers'][i * 2 + 1].get_ydata()[1] - box['whiskers'][i * 2 + 1].get_ydata()[0]) * thre
# 通常的手段是先将异常值设置为缺失值,然后再用处理缺失值的方法完成后续流程
df_data[col[i]] = df_data[col[i]].apply(lambda x: np.nan if (x < lower_bound) | (x > upper_bound) else x)
return df_data.values
threshold = [2.5, 3.0, 3.5]
# threshold = [0.0, 1,0, 2.0]
for thre in threshold:
print('THRESHOLD: %.2f\n' % thre)
## normalize with MinMax and Std on features
features = normalize(data)
thrunk_z_score(f_train[:, 1:-2], thre)
# features[:, 1:-2] = thrunk_box(features[:, 1:-2], thre)
## nan values: 1. fill by mean/mode/knn 2. drop 3. interpolate
# 1. mean/mode/knn
df_features = pd.DataFrame(features)
for col in df_features.columns:
# with mean
temp = df_features[col].copy()
temp = temp.dropna(how='any', axis=0)
df_features[col].fillna(np.random.choice(temp), inplace=True)
features = df_features.values
# # 2. drop
# df = pd.DataFrame(f_train)
# df['fake'] = data['fake'].copy()
# df.dropna(axis=0, how='any', inplace=True)
# f_train, l_train = df.drop(columns=['fake']).values, df.fake.values
## splitting training, validation, testing sets
from sklearn.model_selection import train_test_split
f_train, f_test, l_train, l_test = train_test_split(features, data.fake, test_size=0.3, random_state=0)
# f_train, f_test, l_train, l_test = train_test_split(features[:, :-1], features[:, -1], test_size=0.3, random_state=0)
f_test, f_val, l_test, l_val = train_test_split(f_test, l_test, test_size=0.33, random_state=42)
## Kbin
from sklearn.preprocessing import KBinsDiscretizer
kb = KBinsDiscretizer(n_bins=4, encode='ordinal', strategy='kmeans')
f_train_1 = kb.fit_transform(f_train[:, 1:-2])
f_test_1 = kb.transform(f_test[:, 1:-2])
f_val_1 = kb.transform(f_val[:, 1:-2])
f_train = np.concatenate((f_train[:, 0].reshape(-1, 1), f_train_1, f_train[:, -2:].reshape(-1, 2)), axis=1)
f_test = np.concatenate((f_test[:, 0].reshape(-1, 1), f_test_1, f_test[:, -2:].reshape(-1, 2)), axis=1)
f_val = np.concatenate((f_val[:, 0].reshape(-1, 1), f_val_1, f_val[:, -2:].reshape(-1, 2)), axis=1)
## combination of sampling and self-defined validation set
from sklearn.ensemble import RandomForestClassifier
from sklearn.svm import SVC
under = np.linspace(0.02, 0.05, 7)
over = np.linspace(0.3, 0.9, 7)
params = {'criterion': ['gini', 'entropy'],
'max_depth': [1, 2],
'min_samples_split': [20, 30, 40],
'min_samples_leaf': [15, 20, 30],
'max_leaf_nodes': [10, 20, 30],
'class_weight': [{0:1, 1:1}, {0:1, 1:2}, {0:1, 1:2}],
}
for i in under:
for j in over:
print('Under ratio %.3f, over ratio %.2f' % (i, j))
r_features, r_labels = under_resample(f_train, l_train, i)
r2_features, r2_labels = over_resample(r_features, r_labels, j)
# f_train_val, l_train_val, pfs = my_cv(r2_features, f_val, r2_labels, l_val)
f_train_val, l_train_val, pfs = my_cv(r2_features, np.concatenate((f_test, f_val)), r2_labels, np.concatenate((l_test, l_val)))
best_params_rf = grid_search(RandomForestClassifier(n_estimators=50, n_jobs=-1, random_state=42), f_train_val, l_train_val, params, cv=pfs, scoring='f1')
# best_params_rf = grid_search(SVC(random_state=42), f_train_val, l_train_val, params, cv=pfs, scoring='f1')
print('\n')
print('\n')
这一次结果终于有明显的提升了,第一次提交得分为 0.090,第二次提交结果为 0.09524。However,这已经是最后两次提交机会了,气哭。
比赛结果反思
- 为什么 kaggle 上的分享文章结果可以那么好看?
我觉得可能有两个主要原因:
一是他们研究的问题是金融诈骗,属于区分性质相对较强的行为;而上市公司的财务造假是一群顶尖的人才殚精竭虑的结果,区分性质经过了一重重的粉饰已经淡化了许多。
二是他们的数据量足够大,数据量在机器学习中一直是个关键问题,我没有使用深度学习的模型,也是因为看了篇文章说深度学习在数据量小时容易过拟合。但是,在比赛结束后,我与其他选手一交流,发现好多得分比我高的人都是用深度学习,真是玄学。
- 离群值应该怎么处置/应用?
虽然有好多文章建议说可以把不平衡的数据集当作异常检测来进行,但是也有好多文章说这样的效果远不如有监督学习。我个人的经验是,既然离群值是少数,那么有两种最可能的情况:一是它能明显区分正负样本,二是它确实是正常范围内的极端情况,没啥区分作用。
对应到此次比赛,我观察到的情况是在不同的阈值下,离群值都没有严格与正样本或负样本相关,所以我将它当作普通噪音去除。这也是合理的,既然是造假,那当然是尽可能造得合理些,不太可能出现极端值。
- 在不平衡的数据集上,要如何有效地做特征选择与降维?
许多博客提到要有效地做特征选择,但是没有谁敢担保哪种方法就一定更好,所以只能是逐个尝试,经验只是能把最有可能的范围不断缩小,减少时间成本。
个人的经验:数据观察是必要的步骤,结合专业知识挑选出最有区分潜力的特征;协方差矩阵只能是作为参考,相关性低不代表区分度低,慎用;随机森林+信息增益容易放大头几个特征,需要配合其他方法来比较再挑选;特征选择与降维的不同方法建议逐个尝试。
- 对于不符合正态分布的特征值,应该怎么转换或处理?
思路一:如果都是正值,可以考虑使用对数进行变换;如果有负值,则向右平移再用对数变换——可能需要在平移前先处理下离群值。
思路二:使用 MinMax,Robust,Sigmoid 等其他标准化方法。
以上。
这还是我第一次实战认真处理不平衡的数据集,本来也没奢望能有多好的结果,能有面试资格已经是超出预期。谨以此文当作实战经验积累的见证。希望各路大佬留言交流下更好的思路~(想破头也想不明白第一名是怎么完虐第二名的……)