CURE算法详解
第二十九次写博客,本人数学基础不是太好,如果有幸能得到读者指正,感激不尽,希望能借此机会向大家学习。这一篇作为可伸缩聚类(Scalable Clustering)算法的第二篇,主要是对CURE(Clustering Using Representative)算法进行详细介绍,其他可伸缩聚类算法的链接可以从《可伸缩聚类算法综述(可伸缩聚类算法开篇)》这篇文章中找到。
CURE算法简介
CURE(Clustering Using Representative)采用了一种新型的层次聚类算法,这种算法介于“单链”和“组平均”之间,他克服了这两种层次聚类算法的不足之处,如下图所示分别使用“单链”与“组平均”处理图1(a)中的数据,“单链”会将由低密度带连接的两个簇错误的合并为一个簇(如图1(c)所示),而“组平均”得到的簇偏向于球形,因此会将图中瘦长的簇拆散(如图1(b)所示),而CURE算法可以处理大型数据、离群点和具有非球形大小和非均匀大小的簇的数据。

该算法采用簇中的多个代表点来表示一个簇,首先选择簇中距离质心最远的点做为第一个点,然后依次选择距离已选到的点最远的点,直到选到
个点为止(一般选择
),这些点捕获了簇的形状和大小。然后将这些选取到的点根据参数
(
)向该簇的质心收缩,距离质心越远的点(例如离群点)的收缩程度越大,因此CURE对离群点是不太敏感的,这种方法可以有效的降低离群点带来的不利影响。
在得到上述缩减后的代表点后,两个簇之间的距离就可以定义为这两个簇中距离最近的两个代表点之间的距离,距离的数学定义如下

其中,簇
的代表点集合表示为
,
可以采用
或其他距离度量方式。然后在CURE层次聚类算法的每一步中对距离最近的簇进行合并。可以看到,当
时每个代表点都缩减到了该簇的质心上,这相当于“组平均”层次聚类,而当
时每个代表点不会缩减,因此相当于“单链”层次聚类。
CURE算法的空间复杂度与数据集大小
呈线性关系,而时间复杂度高达
,对于低维数据集该复杂度可以降低到
,因此其时间复杂度不会高于“组平均”层次聚类。但是CURE依旧不能运行于大型数据集,因此与BIRCH中通过汇总的方式来处理该问题不同的是,CURE采用了两种技术来加速聚类,首先在数据集上进行采样,可以证明采用适当的最小随机采样大小可以足够精准的表示簇的几何信息,我们可以采用“切尔霍夫界限”(Chernoff Bounds)来计算这个最小大小,其次,为了进一步提高聚类速度,CURE还将采样得到的点集进行分割,然后在分割后的子集上运行CURE层次聚类,在删除每个子集中的离群点后,再将每个子集中形成的簇进一步CURE层次聚类产生最终的聚类模型。
在得到采样样本子集的聚类信息后,就可以对数据集中剩下的样本点进行划分,这里是跟据每个簇中的代表点进行划分的,而不是像BIRCH中使用簇质心进行划分,具体的方法是找到距离当前样本点最近的代表点,并将该样本点标记为该点所在的簇。
CURE算法流程
CURE算法的基本流程可以表示为下图(图2),首先从原始数据集中随机抽取一部分样本点作为子集,再对该子集进行划分,在这些划分后的集合上运行CURE聚类算法得到每个集合的簇,并删除其中的离群点,然后对这些簇进一步进行CURE层次聚类,并删除其中的离群点,最后对磁盘中剩余的数据集样本点进行划分。

CURE层次聚类算法
CURE层次聚类算法有以下几个重要的特点:可以识别出任意形状的簇(例如椭圆形)、对于离群点的影响具有鲁棒性、具有线性的空间复杂度和
的时间复杂度(低维数据空间,最坏的情况下为
)。注意,这一部分讨论中用到的数据点个数
是指原始数据集的大小,而不是经过采样或分割后的子集大小。
该算法与一般的凝聚层次聚类算法类似,首先将数据集中的每一个样本点作为一个独立的簇,然后迭代的合并当前簇集合中距离最近的一对簇,与第一部分对距离的定义(如式(1))同样,这里将两个簇之间的距离定义为这两个簇中距离最近的两个代表点(缩减后的)之间的距离。算法的细节伪代码如下图(图3)所示,

其中
代表输入的数据集,
是期望的聚类数目,与簇
距离最近的簇被标记为
,可以看出算法中采用了两种数据结构heap和k-d tree,heap中根据每个簇与其最近簇之间的距离进行排序后,对这些簇进行顺序插入,而k-d tree中则存储了每个簇的代表点,它是一种二分查找树,可以对高维数据实现更加有效的存取,在树的每一层根据不同的键值来决定下一步进入到哪个分支中去,例如一个二维数据的k-d tree进行查找时,在根节点测试第一个维度的值,在下一节点中测试第二个维度的值,k-d tree用于簇合并过程(算法第7行)。
下面分别对算法中的每一步进行介绍,
算法第1-2行:分别将两种数据结构heap和k-d tree初始化为
和
;
算法第3行:循环停止条件是判断
中存储的簇个数是否不大于
;
算法第4-8行:从
中提取出(与其最近的簇之间的距离)最小的簇
,然后将距离
最近的簇
从
中移除,将这两个簇合并为
,然后将
和
的代表点从
中移除,并将合并之后的簇
的代表点插入
中;
算法第9行:对距离
最近的簇进行声明,其中簇
是
中的任意一个簇;
算法第10行:对
中的每一个簇
进行迭代;
算法第11-12行:找出距离
最近的簇
;
算法第13-23行:其中包含了两个条件判断,第一个判断与
距离最近的簇(
)是否是
或者
,如果满足这个条件就进入第二个判断,如果不满足就继续判断
与其距离最近的簇(
)之间的距离是否大于
与
的距离,如果满足该条件则将
的最近簇设置为
,并更新
中
的信息,如果也不满足该条件,那么就什么也不做。第二个判断(假设
的最近簇为
)中,如果
与
之间的距离小于
与
之间的距离,就在
中重新寻找一个与
距离最近的簇
,并且要求
与
之间的距离小于
与
之间的距离,如果找不到这样的簇,函数closeset_cluster()返回的是
,当
与
之间的距离大于
与
之间的距离时,将
的最近簇设置为
,完成第二个条件判断之后,就更新
中
的信息;
算法第25行:将
的信息有序的插入到
中。
下面对簇合并过程(图4)进行详细介绍,该过程首先将被合并的两个簇
和
中的样本点合并到簇
中,然后计算
的质心并初始化
的临时代表点集tmpSet,在第一层for循环中依次提取
的代表点并装入tmpSet,第二层for循环是查找代表点的过程,具体方法与文章第一部分介绍的代表点定义相同,这里不做赘述,在第二个for循环中,将tmpSet中的每个点进行收缩得到最终的代表点集
。原文中还提到了一种改进版的簇合并方式,在选择代表点时,查找范围不再是
和
的所有样本点,而是他们各自的代表点所组成的集合,这样做有两个好处,时间复杂度降低为
而且由于这些代表点来自于不同的簇因而得到了相当好的分散。

采样
为了适应大型数据,CURE采用随机抽样将原始数据集中的部分点提取出来,然后试图在这些点上实施CURE层次聚类算法,采样形成的数据子集要适应内存的需要并且与原始数据集相比要足够小。因此,这种随机采样的方法会大大提升CURE的执行速度,并且由于采样过程会对离群点进行过滤因而可以提高聚类质量。
原文推荐了一种随机采样算法《J. Vitter. Random sampling with a reservoir. ACM Zkansactions on Mathematical Software, 11(1):37-57(1985).》,也同时指出可以使用其他的算法进行采样,而且依据过往的经验,这些算法相较于在采样得到的数据集上进行聚类的执行时间要短得多。实际上,由于我们采用原始数据集的随机抽样,必然会有一定的可能性丢失重要的聚类信息,或者造成错误的聚类,这是算法所无法避免的,直到现在依旧没有办法可以同时提高精确度和效率,但是在多次实验中发现,如果选择合适的采样大小,那么最终产生的聚类效果还是很好地,因此原文对采样大小所依照的“切尔霍夫边界”进行了证明和推导,这里只引用其定义式,簇
最适合的采样大小
可以表示为,
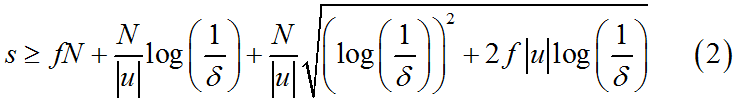
其中,
是采样大小,
是最低采样率,它取决于簇的密度和簇之间的分离度,分离度越高、密度越高,
就设置的越高,
是簇
的大小,
是整个数据集的大小,
是从
个样本点中采样得到
个样本点的概率(
)。
如果两个簇之间的距离非常小,随机采样之后进行聚类可能会使得算法无法区别这两个簇,这时需要将每个簇看做一个个子簇的集合,然后分别在这些子簇上进行采样。
分割
为了进一步提高算法的运行速度,CURE还将抽样得到的数据集进一步分割为 个部分,这样每个部分中的样本点数为 ,然后分别在这些部分进行CURE聚类,每个部分产生 个簇,或者设置一个阈值,如果当前用于合并的两个簇之间的距离大于这个阈值就停止合并。完成聚类后共产生 个簇,然后将这些簇进一步合并,可以证明,这种方法不会降低聚类质量,而且可以大大加快CURE的速度。
离群点的处理
在进行随机采样时,会过滤掉大部分的离群点,此外,在随机采样得到的数据集中存在的少量离群点由于分布在整个原始数据空间,因而被随机采样进一步隔离了。在进行CURE凝聚层次聚类时,需要将每个点单独初始化为一个簇,并将距离最近的点合并为一个簇,由于离群点往往距离样本中的其它点很远,因此他所代表的簇增长的最为缓慢,以至于簇的大小远远小于正常的簇。
以上述讨论作为契机,我们将层次聚类中的离群点识别分为两个阶段,第一个阶段是在聚类算法执行到某一阶段(或称当前的簇总数减小到某个值)时,根据簇的增长速度和簇的大小对离群点进行一次识别,需要注意的是,如果这个阶段选择的较早(即簇总数依旧很大)的话,会将一部分本应被合并的簇识别为离群点,如果这个阶段选择的较晚(即簇总数过少)的话,离群点很可能在被识别之前就已经合并到某些簇中,因此原文推荐当前簇的总数为数据集大小的1/3时,进行离群点的识别。第一阶段有一个很明显的问题,就是当随机采样到的离群点分布的比较近时(即使可能性比较小),这些点会被合并为一个簇,而导致无法将他们识别出来,这时就需要第二阶段的来进行处理。由于离群点占的比重很小,而在层次聚类的最后几步中,每个正常簇的粒度都是非常高的,因此很容易将他们识别出来,一般当簇的总数缩减到大约为
时,进行第二阶段的识别。
参考资料
【1】《机器学习》周志华
【2】《CURE: an efficient clustering algorithm for large databases》SUDIPTO GUHA, RAJEEV RASTOGI, and KYUSEOK SHIM