原创不易,转载前请注明博主的链接地址:Blessy_Zhu https://blog.csdn.net/weixin_42555080
本次代码的环境:
运行平台: Windows
Python版本: Python3.x
IDE: PyCharm

一、k-means算法简介
K-means算法是很典型的基于距离的聚类算法,采用距离 作为相似性的评价指标,即认为两个对象的距离越近,其相似度就越大。该算法认为簇是由距离靠近的对象组成的,因此把得到紧凑且独立的簇作为最终目标。 k-means算法特点在于:同一聚类的簇内的对象相似度较高;而不同聚类的簇内的对象相似度较小
k-means是用来解决著名的聚类问题的最简单的非监督学习算法之一。
该过程遵循一个简易的方式:
- 将一组数据划分为预先设定好的k个簇。其主要思想是为每个簇定义一个质心。设置这些质心需要一些技巧,因为不同的位置会产生不同的聚类结果。因此,较好的选择是使它们互相之间尽可能远
- 接下来将数据中的每个点归类为距它最近的质心,距离的计算可以是欧式距离、曼哈顿距离、切比雪夫距离等。如果所有的数据点都归类完毕,那么第一步就结束了,早期的聚合过程也相应完成。
- 然后,我们根据上一步所产生的结果重新计算k个质心作为各个簇的质心。一旦获得k个新的质心,我们需要重新将数据集中的点与距它最近的新质心进行绑定。一个循环就此产生。
- 作为循环的结果,我们发现k个质心逐步改变它们的位置,直至位置不再发生变化为止。
k-means算法是数值的、非监督的、非确定的、迭代的。
二、k-means算法理解
2.1 经典K-means算法
2.1.1 主要内容
从所有的观测实例中随机取出k个观测点,作为聚类的中心点;然后遍历奇遇的观测点找到各自距离最近的聚类中心点,并将其加入该聚类中。这样,我们便有了一个初始的聚类结果,这是一次迭代过程。
2、每个聚类中心都至少有一个观测实例,这样,我们便可以求出每个聚类的中心点,作为新的聚类中心(该聚类中所有实例的均值),然后再遍历其他所有的观测点,找到距离其最近的中心点,并加入到该聚类中。
3、如此重复步骤2,直到前后两次迭代得到的聚类中心点不再发生变化为止。
该算法旨在最小化一个目标函数——误差平方函数(所有的观测点与其中心点的距离之和)。

可见如下的迭代过程:

它的特点是:
- 初始质心点通常是随机选取的
- 不同的初始质心可能产生的聚类不同
- 质心是一个簇中点的平均值
- 度量使用Euclidean欧几里得距离, cosine 相似度量, 相关性度量等.
- 对上述一般的相似性度量,K-means都会收敛
- 大多数经过前几次迭代就能收敛
- 通常终止条件是‘簇中的点变化很少时’
- 时间复杂度为 O( nKId )
- n = 点的个数, K = 聚类的个数,
- I = 迭代次数, d = 属性个数
2.1.2 度量方法
- 最常用的度量时误差的平方和(Sum of Squared Error, SSE)
对于每个点, 其误差是到最近质心的距离
为了得到 SSE, 我们对每个点的误差求平方和
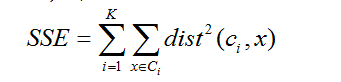
x是簇 Ci 的点, ci 是簇Ci的代表点
可以证明 ci 对应于簇的质心(均值)
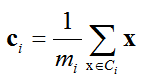
给定两个聚类, 我们可以选择具有最小SSE的聚类 - K-means也可以用于非欧氏空间数据
例, 文档数据 通常, 文档用余弦相似性度量
距离是相异性度量, 而余弦是相似性度量
质心仍然用均值
最近的质心是最相似的质心
目标: 最大化簇中文档与簇的质心的相似性
总凝聚度(total cohesion)
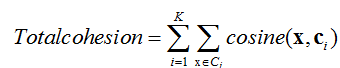
- K均值:邻近度、质心和目标函数的常见选择

2.1.3 初始聚类中心问题解决方法
- 1 多次运行
多次运行,每次选择不同的初始质心,聚类效果和数据集、k有关,不一定达到好的效果 - 2 取样并使用层次聚类技术对它聚类,从层次聚类中提取k个簇,并用这些簇的质心作为初始质心(在下列情况下有效:样本相对小、k相对与样本较小)
- 3 选择分离的质心:即随机选取第一个质心,后面的质心选取应和已经选取的质心距离最远
- 可能选在非稠密区
- 4 后处理:对聚类结果进行处理
- 5 使用二分 K-means,它用于对初始化问题不敏感的情况。
K-means 的变种,可以产生划分或层次聚类
先将所有的点分裂两个簇,再选择一个继续分裂,直到产生k个簇

- 6 增量更新质心
可以在点到簇的每次指派之后,增量地更新质心,而不是在所有的点都指派到簇中之后才更新族质心注意,每步需要零次或两次簇质心更新,因为一个点或者转移到一个新的簇(两次更新),或者留在它的当前簇(零次更新)。使用增量更新策略确保不会产生空簇,因为所有的簇都从单个点开始:并且如果一个簇只有单个点,则该点总是被重新指派到相同的簇。
此外,如果使用增量更新,则可以调整点的相对权值;例如,点的权值通常随聚类的进行而减小。尽管这可能导致更好的准确率和更快的收敛性,但是在千变万化的情况下,选择好的相对权值可能是困难的。这些更新问题类似于人工神经网络的权值更新。
增量更新的另一个优点是使用不同于“最小化SSE"”的目标。假设给定一个度量簇集的目标函数。当我们处理一个 点时,我们可以对每个可能的簇指派计算目标函数的值,然后选择优化目标的族指派。
缺点方面,增量地更新质心可能导致次序依赖性。换言之,所产生的簇可能依赖于点的处理次序。尽管随机地选择点的处理次序可以解决该问题,但是,基本K均值方法在把所有点指派到族中之后才更新质心并没有次序依赖性。此外,增量更新的开销也稍微大;一些。然而,K均值收敛相当快,因此切换簇的点数很快就变得相对较小。
使算法对序的受化有和
2.1.4 K-means的局限性
当聚类的大小、密度、形状不同时,K-means 聚类的结果不理想
数据集包含离群点时,K-means 聚类结果不理想
两个类距离较近时,聚类结果不合理




2.2 改进的K-means算法
上面介绍的k-means 算法是一种非常简单并且使用广泛的聚类算法,但是
- k 值需要预先给定,很多情况下 k 值的估计很困难。
- K-Means 算法对初始选取的聚类中心点很敏感,不同的中心点聚类结果有很大的不同。也就是说,有可能陷入局部最优解。
- 对离群点敏感,聚类结果易产生误差。
- 相似性度量的函数不同也会对聚类结果产生影响。
接下来针对 k-means 的缺陷,总结对k-means的改进。从初始中心点的选取、离群点的检测与去除、相似性度量等几个方面进行概括、比较。
2.2.1 距离和相似性度量
传统的 k-means 算法使用欧几里得距离来度量相似度。
改进的措施是:
- 采用了欧式距离、平方欧式距离、曼哈顿距离、余弦距离、谷本距离分别作为相似度度量对文本数据进行处理,实验结果显示余弦距离、谷本距离者在文本聚类中的表现更优。不同的测度距离作为相似性度量对聚类结果会产生不同的影响,对于不同类型的数据也应采用不同的距离函数作为相似度度量。
- 针对 k-means 算法不能求解非线性流形聚类的缺陷,用空间密度相似性度量来代替欧几里得距离,使 k-means算法能够适应数据集的分布。同一簇中的数据点应位于高密度区域,不同簇中的数据点应该用低密度区域分隔开来。所以需要压缩高密度区域的距离,放大低密度区域的距离。
- 针对比例数据,提出用 Aitchison 距离度量来对比例数据进行聚类。使用 Aitchison 距离、欧几里德对数距离、余弦距离、Kullback 距离、Matisuita 距离进行了对比实验,聚类结果显示 Aitchison 距离度量最适合所有,因为较高的轮廓值,聚类更合适。对于图像比例数据聚类,使用 Aitchison 距离作为初始化步骤可以提供适用于比例数据的更好的混合结果。
2.2.2 初始聚类中心的选取
初始聚类中心的选取对 k-means 算法聚类结果的影响很大,不同的初始聚类中心,可能会产生不同的聚类结果。也可以说,k-means算法是一种贪心算法,容易陷入局部最优解。解决办法如下:
- 类簇中心都处在局部密度比较大的位置,且距离具有比它更大的局部密度的对象相对较远的思想。运用此思想可以确定最佳初始聚类中心及数据集的最佳类簇数目。在这个思想的基础上,为了避免密度对截断距离的依赖性,重新定义了计算样本局部密度 ρ i 的方法,计算样本点到具有比它更高的局部密度数据对象的最近距离 δ i (当样本点 x i 是数据集中具有最大局部密度的样本点时,δ i 定义为 x i 和距离他最远的样本点之间的欧氏距离)。根据 ρ i 和 δ i 构造决策图,运用统计学中残差分析的方法,选取残差绝对值大于阈值的异常点,即为聚类中心。在二维以及高维数据集上的实验结果均验证了该算法的有效性。但是不足之处在于这个算法适用于比较集中的数据集,稀疏的数据集结果并不理想。
- 采用减法聚类的算法确定初始聚类中心。首先是计算每个样本点的山峰函数值,选取山峰函数值最大的点作为聚类中心。选取下一个聚类中心时要消除已经确定的聚类中心的影响,就修改山峰函数,减去上一个确定的聚类中心的比例高斯函数,如此反复,直到得到足够多的聚类中心。这个方法的缺点在于对于离群点、异常值抗干扰能力比较弱,且需要设置的参数较多(一般需要 3 个)。
- 采用了四分位数的概念来确定初始聚类中心。首先采用特征选择的方法选取数据有意义的属性。然后将将属性的值按照顺序排列,以分成两类为例,将数据集的上四分位点和下四分位点作为初始聚类中心,计算所有样本点到到这两个聚类中心的距离,进行分类;接下来更新聚类中心,将每类所有样本点的均值作为新的聚类中心,直到类簇不再发生变化。这个方法不足之处是当数据集比较大时,花费时间会比较长。
- 采用最大 - 最小准则算法初步确定初始聚类中心,然后通过FLANN(快速最近邻搜索库)将聚类中心偏移到尽可能地靠近实际的聚类中心。最大 - 最小准则算法是首先随机选取一个点作为第一个聚类中心,选取距离这个点最远的点作为第二个聚类中心,然后计算每个点到这两个聚类中心的距离,选取较小的距离加入到集合 V 中,在集合 V 中选取距离最远的点作为下一个聚类中心,依次类推,直到最大最小距离不大于θ•D 1,2 (D 1,2 为第一个和第二个聚类中心的距离)。 FLANN 是一个在高维空间中快速搜索k 个最近邻居的库。使用 FLANN 找到聚类中心的 k 近邻,计算中心点即为新的聚类中心。
2.2.3离群点的检测
K-means 算法对于离群点敏感,对聚类结果产生很大的影响,因此离群点的检测与删除极为重要。解决的方法有:
- 基于密度的方法是一种流行的异常值检测方法。 它计算每个点的局部离群因子(LOF)。 一个点的 LOF 是基于该点附近区域的密度和它的邻居的局部密度的比值。LOF 方法依赖于用户提供的最小数量点,用于确定邻居的大小。
- 建立了一个基于本地距离的离群因子(LDOF)来测量分散的数据集对象的离群程度。LDOF 使用一个对象到它的邻居的相对位置,以确定物体偏离其邻近区域的程度。为了方便实际应用中的参数设置,在离群值检测方法中使用了一种 top-n 技术,其中只有具有最高值的对象才被视为离群值。与传统的方法(如前 n 和顶 n)相比,方法是在分散的数据中检测出离群值。
- 通过添加上限范数和一种有效的迭代重加权算法,来减小离群点的影响。离群点的检测发生在每次聚类中心迭代时,每个样本点到聚类中心的距离大于给定的参数 ε,便会被去除。并且重新给样本分配权重,低错误率的样本具有更高的权重。这个方面的缺陷在于参数 ε 需要人为的设置与调整,不同的ε值导致的聚类结果准确率不同。
- 提出了k-means-sharp 算法,通过从点到质心距离的分布得到的全局阈值自动检测离群点。离群点检测过程和聚类过程同时完成。假设 k-means的数据呈高斯分布,离群点检测发生在每次聚类中心更新时,计算每个样本点到聚类中心的距离,如果距离大于 3σ,则为离群点,其中σ=1.4826MADe, MADe 为每个点到中值点的距离组成的所有数据的中值点。因此,每次更新聚类中心时,就会去除一部分离群点。
2.2.4 k-means算法的其他改进
最近几年出现了遗传算法、粒子群算法、萤火虫算法、蚁群算法等与传统的 kmeans 算法相结合的改进算法,这几类算法的共同点是具有一定的全局优化能力,理论上可以在一定的时间内找到最优解或近似最优解。通常是使用这些算法来寻找 k-means 算法的初始聚类中心。
- 将k-means和遗传算法结合的 k-means 算法优于传统的 k-means 算法。遗传 k-means 算法就是把每个聚类中心坐标当成染色体基因。聚类中心个数就是染色体长度,对若干相异染色体进行初始化操作并将其当成一个种群进行遗传操作,最终获得适应度最大染色体,而最优聚类中心坐标就是解析出的各中心点坐标。
- 将粒子群算法与 k-means算法结合,多子群多于多子群粒子群伪均值(PK-means)聚类算法,理论分析和实验表明,该算法不但可以防止空类出现,而且同时还具有非常好的全局收敛性和局部寻优能力,并且在孤立点问题的处理上也具有很好的效果。
- 基于萤火虫优化的加权 K-means 算法。该算法在提升聚类性能的同时,有效增强了算法的收敛速度。在实验阶段,通过 UCI 数据集中的几组数据对该算法进行了聚类实验及有效性测试,实验结果充分表明了该算法的有效性及优越性。可见,将k-means算法与其他算法相结合,可以弥补 k-means 算法的不足,获得更好的聚类效果。
三、基于k-means算法的iris数据集Python实现
3.1 iris数据集可视化
from sklearn import datasets
iris = datasets.load_iris()
X, y = iris.data, iris.target
data = X[:,[1,3]] # 为了便于可视化,只取两个维度
plt.scatter(data[:,0],data[:,1]);
plt.show()

3.2 计算欧式距离
需要为每个点找到离其最近的质心,需要用这个辅助函数。
def distance(p1,p2):
"""
Return Eclud distance between two points.
p1 = np.array([0,0]), p2 = np.array([1,1]) => 1.414
"""
tmp = np.sum((p1-p2)**2)
return np.sqrt(tmp)
distance(np.array([0,0]),np.array([1,1]))
1.4142135623730951
3.3 随机质心
在给定数据范围内随机产生k个簇心,作为初始的簇。随机数都在给定数据的范围之内dmin + (dmax - dmin) * np.random.rand(k)实现。
def rand_center(data,k):
"""Generate k center within the range of data set."""
n = data.shape[1] # features
centroids = np.zeros((k,n)) # init with (0,0)....
for i in range(n):
dmin, dmax = np.min(data[:,i]), np.max(data[:,i])
centroids[:,i] = dmin + (dmax - dmin) * np.random.rand(k)
return centroids
centroids = rand_center(data,2)
centroids
array([[ 2.15198267, 2.42476808],
[ 2.77985426, 0.57839675]])
3.4 k均值聚类
这个基本的算法只需要明白两点。
- 给定一组质心,则簇更新,所有的点被分配到离其最近的质心中。
- 给定k簇,则质心更新,所有的质心用其簇的均值替换
当簇不在有更新的时候,迭代停止。当然kmeans有个缺点,就是可能陷入局部最小值,有改进的方法,比如二分k均值,当然也可以多计算几次,去效果好的结果。
def kmeans(data,k=2):
def _distance(p1,p2):
"""
Return Eclud distance between two points.
p1 = np.array([0,0]), p2 = np.array([1,1]) => 1.414
"""
tmp = np.sum((p1-p2)**2)
return np.sqrt(tmp)
def _rand_center(data,k):
"""Generate k center within the range of data set."""
n = data.shape[1] # features
centroids = np.zeros((k,n)) # init with (0,0)....
for i in range(n):
dmin, dmax = np.min(data[:,i]), np.max(data[:,i])
centroids[:,i] = dmin + (dmax - dmin) * np.random.rand(k)
return centroids
def _converged(centroids1, centroids2):
# if centroids not changed, we say 'converged'
set1 = set([tuple(c) for c in centroids1])
set2 = set([tuple(c) for c in centroids2])
return (set1 == set2)
n = data.shape[0] # number of entries
centroids = _rand_center(data,k)
label = np.zeros(n,dtype=np.int) # track the nearest centroid
assement = np.zeros(n) # for the assement of our model
converged = False
while not converged:
old_centroids = np.copy(centroids)
for i in range(n):
# determine the nearest centroid and track it with label
min_dist, min_index = np.inf, -1
for j in range(k):
dist = _distance(data[i],centroids[j])
if dist < min_dist:
min_dist, min_index = dist, j
label[i] = j
assement[i] = _distance(data[i],centroids[label[i]])**2
# update centroid
for m in range(k):
centroids[m] = np.mean(data[label==m],axis=0)
converged = _converged(old_centroids,centroids)
return centroids, label, np.sum(assement)
3.5 重复运行取最优
best_assement = np.inf
best_centroids = None
best_label = None
for i in range(10):
centroids, label, assement = kmeans(data,2)
if assement < best_assement:
best_assement = assement
best_centroids = centroids
best_label = label
data0 = data[best_label==0]
data1 = data[best_label==1]
3.6 结果可视化
把数据分为两簇,绿色的点是每个簇的质心,从图示效果看,聚类效果还不错。
fig, (ax1,ax2) = plt.subplots(1,2,figsize=(12,5))
ax1.scatter(data[:,0],data[:,1],c='c',s=30,marker='o')
ax2.scatter(data0[:,0],data0[:,1],c='r')
ax2.scatter(data1[:,0],data1[:,1],c='c')
ax2.scatter(centroids[:,0],centroids[:,1],c='b',s=120,marker='o')
plt.show()

四 总结
K-means 的发展已经经历了很长的一段时间,它所具有的独特优势使得其被广大研究者不断地优化和使用。本文对 k-means 进行简单介绍。同时对 k-means 的研究和改进还应注意以下几点:
- (1)随着互联网技术的发展,数据量呈现出一种指数级增长。如何高效地对这些海量数据进行处理和分析已成为当前研究热点。传统的 k-means 算法难以有效处理大的数据集。所以将并行计算和 k-means 算法结合,并不断地对其加以改进和优化,是处理海量数据的有效途径。
- (2)k-means 聚类算法的改进有许多依然需要用户输入参数,传统的 k-means 算法的k 值的确定研究不多。所以参数的自确定是一个不断需要发展研究的问题。
- (3)从文中可以看出,现在存在着各种各样的数据类型,文本、图像、混合型数据等等,现有的多是处理二维数据,对高维数据处理的研究不多,需要对 k-means 算法进行扩展,以得到一个能够适应大多数类型数据类型的 k-means 算法模型。
这篇文章就到这里了,欢迎大佬们多批评指正,也欢迎大家积极评论多多交流。

参考文章
【1】Kmeans算法的Python实现
【2】k-means聚类的传统算法和优化
【3】k-means算法、性能及优化
【4】论文:K-means 算法研究综述
附完整代码
from sklearn import datasets
import matplotlib.pyplot as plt
import numpy as np
iris = datasets.load_iris()
X, y = iris.data, iris.target
data = X[:,[1,3]] # 为了便于可视化,只取两个维度
def kmeans(data, k=2):
def _distance(p1, p2):
"""
Return Eclud distance between two points.
p1 = np.array([0,0]), p2 = np.array([1,1]) => 1.414
"""
tmp = np.sum((p1 - p2) ** 2)
return np.sqrt(tmp)
def _rand_center(data, k):
"""Generate k center within the range of data set."""
n = data.shape[1] # features
centroids = np.zeros((k, n)) # init with (0,0)....
for i in range(n):
dmin, dmax = np.min(data[:, i]), np.max(data[:, i])
centroids[:, i] = dmin + (dmax - dmin) * np.random.rand(k)
return centroids
def _converged(centroids1, centroids2):
# if centroids not changed, we say 'converged'
set1 = set([tuple(c) for c in centroids1])
set2 = set([tuple(c) for c in centroids2])
return (set1 == set2)
n = data.shape[0] # number of entries
centroids = _rand_center(data, k)
label = np.zeros(n, dtype=np.int) # track the nearest centroid
assement = np.zeros(n) # for the assement of our model
converged = False
while not converged:
old_centroids = np.copy(centroids)
for i in range(n):
# determine the nearest centroid and track it with label
min_dist, min_index = np.inf, -1
for j in range(k):
dist = _distance(data[i], centroids[j])
if dist < min_dist:
min_dist, min_index = dist, j
label[i] = j
assement[i] = _distance(data[i], centroids[label[i]]) ** 2
for m in range(k):
centroids[m] = np.mean(data[label == m], axis=0)
converged = _converged(old_centroids, centroids)
return centroids, label, np.sum(assement)
best_assement = np.inf
best_centroids = None
best_label = None
for i in range(10):
centroids, label, assement = kmeans(data,2)
if assement < best_assement:
best_assement = assement
best_centroids = centroids
best_label = label
data0 = data[best_label==0]
data1 = data[best_label==1]
fig, (ax1,ax2) = plt.subplots(1,2,figsize=(12,5))
ax1.scatter(data[:,0],data[:,1],c='c',s=30,marker='o')
ax2.scatter(data0[:,0],data0[:,1],c='r')
ax2.scatter(data1[:,0],data1[:,1],c='c')
ax2.scatter(centroids[:,0],centroids[:,1],c='b',s=120,marker='o')
plt.show()