简介
外显子测序是指利用序列捕获技术将全基因组外显子区域DNA捕捉并富集后进行高通量测序的基因组分析方法。全外显子组测序仅需对全基因组的2%左右的区域进行测序,就能够得到超过95%的已知功能区域的信息,而这些区域涵盖了95%的孟德尔疾病致病突变以及多种疾病易感多态性位点。因此,全外显子组测序已经成为当前疾病相关基因检测的常用方法,广泛应用于寻找复杂疾病的致病基因和易感基因等。下面对外显子组测序数据的分析流程进行简要的介绍,分析流程见下图。
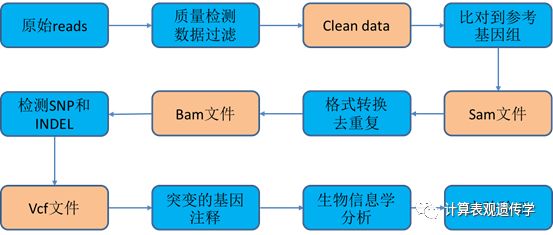
其中,数据的质量检查通常使用Fastqc软件完成,比对软件通常使用BWA,该软件是基于Burrows Wheeler转换法,对参考基因组进行压缩并建立索引,再进行比对,再通过查找和回溯来定位。数据排序,去重复通常使用samtools软件将比对后的sam文件转化为二进制的bam文件,然后使用Picardtools软件对bam文件排序,最后使用Picard-toolkit软件的MarkDuplicates工具去除冗余数据。通常使用samtools软件和VarScan软件检测SNP和INDEL,输出结果为vcf格式文件,其中包括变异信息和与dbsnp数据库比对的信息。变异的基因注释通常使用annovar软件,注释内容通常包括突变位置、突变分类(杂合或纯合)、基因名、转录本名、外显子号、蛋白突变、氨基酸突变、rs号、1000Genome基因组频率、功能预测模型(SIFT、polyphen)、数据库(cosmic70、clinvar)等。最后可以使用生物信息学分析方法找到疾病的致病候选基因变异,再经过大样本量的实验验证,找到真正的致病突变。
参考文献:
《高通量测序与高性能计算理论和实践》陈禹保 黄劲松
往期「精彩内容」,点击回顾
DNA测序历史 | CircRNA数据库 | Epigenie表观综合 | 癌症定位
BWA介绍 | 源码安装R包 | CancerLocator | lme4 | 450K分析
乳腺癌异质性 | BS-Seq | 隐马模型 | Circos安装 | Circos画图
KEGG标记基因 | GDSC | Meta分析 | R线性回归和相关矩阵
精彩会议及课程,点击回顾