天然产物在药物发现的历史上占有非常重要的地位,许多药物仍然是天然产物及其衍生物。因此,过去大型制药公司都拥有“天然产物化学”部门,但是低通量天然产物的筛选逐渐变得无利可图并被关闭。
与药物发现化学中通常合成的化合物相比,天然产物的结构特征为:
- 许多稠密的环结构
- 许多不对称中心
- 芳香环更少,sp3碳更多
- 氧原子百分比高,氮原子百分比低
天然产物化合物的特征
为了将“ 天然产物相似性 ”用作库设计的指标,有必要了解天然产物化合物的特征。与合成化合物相比,天然产物的结构如前所述:
- 许多稠密的环结构
- 许多不对称中心
- 芳香环更少,sp3碳更多
- 氧原子百分比高,氮原子百分比低
作为理化参数:
- 高脂肪溶解度
- 高极性表面积
此外,作为合成库的指南,重点关注天然产物中经常出现的结构多样性和骨架,Schreiber倡导的“ 多样性导向的合成:DOS ”和Waldmann 倡导的“ 生物导向的合成:BIOS ”等。是众所周知的。
Natural Product-likeness (NP-likeness)
论文[Natural Product-likeness Score and Its Application for Prioritization of Compound Libraries]的作者试图将化合物和天然产物在化学空间中的接近度评分为NP-likeness,可以说它drug-likeness。
根据某些规则将生成的结构片段化,并通过对天然产物(NP)中经常发现的位点和合成化合物(SM)中经常发现的位点进行加权来对每个片段进行评分。基本思想就像两类分类问题,当给定化合物时,该问题将返回“天然产物”或“合成产物”。
NP相似性得分介于-5到5之间,数字越大,则其越有可能是天然产物。
RDKit-NP_Score
从rdkit / Contrib / NP_Score /(https://github.com/rdkit/rdkit/tree/master/Contrib/NP_Score)获取必要的脚本。
导入库
from rdkit import rdBase, Chem
from rdkit.Chem import Descriptors
from rdkit.Chem import AllChem, Draw, PandasTools
import itertools
import matplotlib as mpl
from matplotlib import style
import matplotlib.pyplot as plt
%matplotlib inline载入数据
df = PandasTools.LoadSDF('naturalproduct_455cmpd.sdf')
df.head()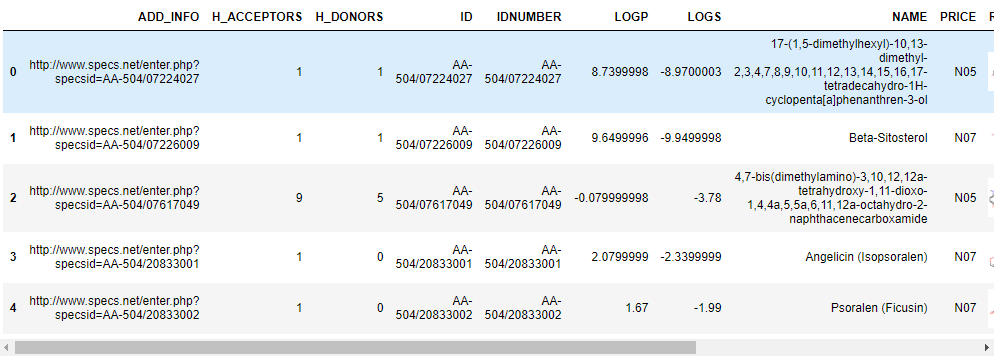
绘制分子
PandasTools.FrameToGridImage(df[:8], column='ROMol', legendsCol='ID', molsPerRow=4, subImgSize=(300,300))
导入模型
import pickle
with open('publicnp.model', 'rb') as f:
fs = pickle.load(f)
import npscorer
fs_scores = [npscorer.scoreMol(m, fs) for m in df.ROMol]
df['np-likeness'] = fs_scores
with mpl.style.context('seaborn'):
plt.hist(fs_scores, density=True, bins=range(-5,6))
plt.xlabel('NP-likeness score')
plt.ylabel('rel freq')
df.ROMol[df['np-likeness'].idxmax()]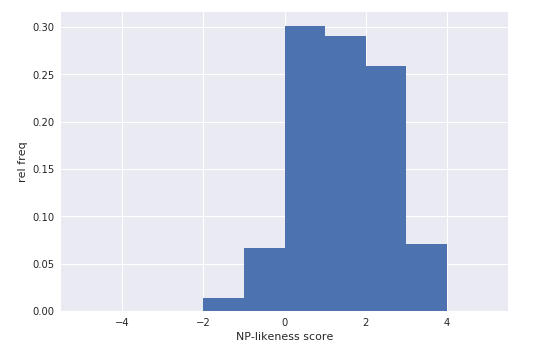
描述符计算,计算NP Score按阳性/阴性分类,为2x2网格中的每个描述符创建直方图。
df['MolLogP'] = df.ROMol.map(Descriptors.MolLogP)
df['MW'] = df.ROMol.map(Descriptors.MolWt)
df['TPSA'] = df.ROMol.map(Descriptors.TPSA)
df['Fsp3'] = df.ROMol.map(Descriptors.FractionCSP3)
df_np = df[df['np-likeness'] >= 0]
df_sm = df[df['np-likeness'] < 0]
len(df_np) ### 100
descs = ['MW', 'MolLogP', 'TPSA', 'Fsp3']
with mpl.style.context('seaborn'):
fig, ax = plt.subplots(2,2, figsize=(16,8))
for (i,j), desc in zip(itertools.product(range(2), range(2)), descs):
ax[i,j].hist([df_np[desc], df_sm[desc]], density=True, label=['NP', 'SM'])
ax[i,j].set_xlabel(desc)
ax[i,j].legend(loc='best')
